老实说,模块其实是一个很讨厌的东西,但是它也实实在在的节省了我们很多时间,也符合我的理念:避免重复造轮子!此教程可能过期了,请直接看最新版(perl模块安装大全)
1,perl的那些模块
如果有root权限,用root权限
进入cpan然后install ExtUtils::Installed模块
这样就可以执行instmodsh这个脚本了,可以查看当前环境下所有的模块 Continue reading
老实说,模块其实是一个很讨厌的东西,但是它也实实在在的节省了我们很多时间,也符合我的理念:避免重复造轮子!此教程可能过期了,请直接看最新版(perl模块安装大全)
如果有root权限,用root权限
进入cpan然后install ExtUtils::Installed模块
这样就可以执行instmodsh这个脚本了,可以查看当前环境下所有的模块 Continue reading
此教程可能过期了,请直接看最新版(perl模块安装大全)
发现自己搞服务器遇到的困难还是蛮多的,所以记录了一下,给菜鸟们指个路。
ubuntu对生信菜鸟来说是最好用的linux服务器,没有之一,因为它有apt-get。
前面我简单写了一个perl的cpan安装模块,但是前些天突然发现有些perl模块在cpan里面找不到,所以又总结了一下不同的perl模块安装方法。 Continue reading
发现自己搞服务器遇到的困难还是蛮多的,所以记录了一下,给菜鸟们指个路。
ubuntu对生信菜鸟来说是最好用的linux服务器,没有之一,因为它有apt-get。
理论上perl是不需要更新,但是我就不巧碰到了这个情况,所以也记录一下
linux下升级系统默认安装的perl版本,不建议先rm
先下载tar.gz ...然後手动安装..default 安装到/usr/local/目录下..
然後修改/usr/bin/perl的symbolic link到/usr/local/bin/perl
下载方式不用说了吧,各显神通,笔者习惯用wget.
所以wget[url]http://www.cpan.org/src/perl-5.10.0.tar.gz[/url] .现在最新是5.20
下载完以后解压安装
#tar zxvf perl-5.10.0.tar.gz
#cd perl-5.10.0
#./Configure -des -Dprefix=/usr/local/perl
参数-Dprefix指定安装目录为/usr/local/perl
#make
#make test
#make install
如果这个过程没有错误的话,那么恭喜你安装完成了.是不是很简单?
接下来替换系统原有的perl,有最新的了咱就用嘛.
#mv /usr/bin/perl/ usr/bin/perl.bak
#ln -s /usr/local/perl/bin/perl/ usr/bin/perl
#perl –v
然后就可以了用它来安装一些其它你需要的perl模块了
#perl -MCPAN-e shell
第一次执行的话,会提示安装cpan并要求连接网络下载最新的模块列表.然后就可以安装东西了
cpan[1]> install DBI
之所以要讲一下这个,是因为trinity这个软件居然需要perl的模块才能使用,所以我必须自己安装几个模块。此教程可能过期了,请直接看最新版(perl模块安装大全)
前提是要有root权限,否则只能自己下载perl模块自己解压安装了。 Continue reading
查询需要根据前面建立的索引来做。
这是一个比较复杂的过程,我也是看了bowtie的作者的ppt才慢慢弄懂的,感觉自己也不可能三言两语就说清楚,一般都是辅助图片,动画,再经过多方交流才能慢慢理解。
所以大家呢,就自己去看ppt,看懂那个查询算法。(ppt及代码在我的群里面有共享,欢迎大家加群交流)
这里我简单讲讲我的程序
首先读取索引文件,统计好A,C,G,T的总数
然后把查询序列从最后一个字符往前面回溯。
我创建了一个子函数,专门来处理回溯的问题
每次接受四个参数(左右两端的碱基,上下的阈值),并返回两个参数(新的上下两个阈值)
大家要看懂阈值是如何更新迭代,这样动态的一个个回溯字符串,一个个迭代阈值。
直到四种临界情况的出现。
第一是上下阈值已经相等了,但是我们还没有回溯完全,那就说明字符串只能查找后几个字符,前面还有字符是无法匹配的
第二种情况是上下阈值已经相等了,正巧我们也回溯到了最后一个字符串,那么我们就找到了精确匹配。
第三种情况是已经进行到了最后一个字符串,但是上下阈值还有差值,那么就找到了多个精确匹配点。
最后一种情况是各种非法字符。
然后我简单的测序了一下在病毒的5K基因组里面的精确匹配情况,好像效果还挺好的
但是在酵母里面还有一个问题没有解决,就是取前二十个字符串排序的问题,不够精确,需要重新审视排序结果进行局部优化,可能是需要用堆排序发,具体我还得考虑一个星期,只能等下周上课再看看了,平时太忙了,基本没时间码代码。
这里贴上我的代码给大家看看,
[perl]
$a='CGCTATGTACTGGATGCGCTGGCAAACGAGCCTGCCGTAAG';
while(<>){
chomp;
@F=split;
$hash_count_atcg{$F[0]}++;
$hash{$.}=$_;
}
$all_a=$hash_count_atcg{'A'};
$all_c=$hash_count_atcg{'C'};
$all_g=$hash_count_atcg{'G'};
$all_t=$hash_count_atcg{'T'};
#print "$all_a\t$all_c\t$all_g\t$all_t\n";
$len_a=length $a;
$end_a=$len_a-1;
print "your query is $a\n";
print "and the length of your query is $len_a \n";
foreach (reverse (0..$end_a)){
$after=substr($a,$_,1);
$before=substr($a,$_-1,1);
#对第一个字符进行找阈值的时候,我们需要人为的定义起始点!
if($_ == $end_a){
if ($after eq 'A') {
$start=1;
$end=$all_a;
}
elsif ($after eq 'C') {
$start=$all_a+1;
$end=$all_a+$all_c;
}
elsif ($after eq 'G') {
$start=$all_a+$all_c+1;
$end=$all_a+$all_c+$all_g;
}
elsif ($after eq 'T'){
$start=$all_a+$all_c+$all_g+1;
$end=$all_a+$all_c+$all_g+$all_t;
}
else {print "error !!! we just need A T C G !!!\n";exit;}
}
#如果阈值已经无法继续分割,但是字符串还未查询完
if ($_ > 0 && $start == $end) {
$find_char=substr($a,$_);
$find_len=length $find_char;
#这里需要修改,但是不影响完全匹配了
print "we can just find the last $find_len char ,and it is $find_char \n";
exit;
}
#如果进行到了最后一个字符
if ($_ == 0) {
if ($start == $end) {
print "It is just one perfect match ! \n";
my @F_start=split/\s+/,$hash{$start};
print "The index is $F_start[1]\n";
exit;
}
else {
print "we find more than one perfect match!!!\n";
#print "$start\t$end\n";
foreach ($start..$end) {
my @F_start=split/\s+/,$hash{$_};
print "One of the index is $F_start[1]\n";
}
exit;
}
}
($start,$end)=&find_level($after,$before,$start,$end);
}
sub find_level{
my($after,$before,$start,$end)=@_;
my @F_start=split/\s+/,$hash{$start};
my @F_end=split/\s+/,$hash{$end};
if ($before eq 'A') {
return ($F_start[2],$F_end[2]);
}
elsif ($before eq 'C') {
return ($all_a+$F_start[3],$all_a+$F_end[3]);
}
elsif ($before eq 'G') {
return ($all_a+$all_c+$F_start[4],$all_a+$all_c+$F_end[4]);
}
elsif ($before eq 'T') {
return ($all_a+$all_c+$all_g+$F_start[5],$all_a+$all_c+$all_g+$F_end[5]);
}
else {print "sorry , I can't find the right match!!!\n";}
}
#perl -alne '{next if />/;$all.=$_;}END{print substr($all,308,10)}' lambda_virus.fa
[/perl]
其中第一讲我提到了一个简单的索引产生方式,因为是课堂就半个小时想的,很多细节没有考虑到,对病毒那种几K大小的基因组来说是很简单的,速度也非常快,但是我测试了一下酵母,却发现好几个小时都没有结果,我只好kill掉重新改写算法,我发现之前的测序最大的问题在于没有立即substr函数的实现方式,把一个5M的字符串不停的截取首尾字符串好像是一个非常慢的方式。
所以我优化了那个字符串的函数,虽然代码量变多了,实现过程也繁琐了一点,但是速度提升了几千倍。
time perl bwt_new_index.pl e-coli.fa >e-coli.index
测试了一下我的脚本,对酵母这样的5M的基因组,索引耗费时间是43秒
real 0m43.071s
user 0m41.277s
sys 0m1.779s
输出的index矩阵如下,我简单的截取头尾各10行给大家看,一点要看懂这个index。
首先第一列就是我们的BWT向量,也就是BWT变换后的尾字符
第二列是之前的顺序被BWT变换后的首字符排序后的打乱的顺序。
第三,四,五,六列分别是A,C,G,T的计数,就是在当行之前累积出现的A,C,G,T的数量,是对第一列的统计。
这个索引文件将会用于下一步的查询,这里贴上我新的索引代码,查询见下一篇文章
[perl]
while (<>){
next if />/;
chomp;
$a.=$_;
}
$len=length $a;
open FH_F,">tmp_forward.txt";
open FH_R,">tmp_reverse.txt";
for(my $i=0;$i<=$len-1;$i+=20){
print FH_F substr($a,$i,20);
print FH_F "\n";
}
$rev_a=reverse $a;
for(my $i=0;$i<=$len-1;$i+=20){
print FH_R substr($rev_a,$i,20);
print FH_R "\n";
}
close FH_F;
close FH_R;
$a='';
open FH_F,"tmp_forward.txt";
open FH_R,"tmp_reverse.txt";
#把前一行的所有20bp碱基当做后一行的头部信息
$residue_F=<FH_F>;
$residue_R=<FH_R>;
$i=0;
while ($F_reads=<FH_F>){
$R_reads=<FH_R>;
$F_merge=$residue_F.$F_reads;
$R_merge=$residue_R.$R_reads;
#这样每次就需要处理20个碱基
foreach (0..19) {
$up =substr($F_merge,$_,20);
$down=substr($R_merge,$_,1);
$hash{"$up\t$down"}=$i;
$i++;
}
#处理完毕之后再保存当行的20bp碱基做下一行的头部信息
$residue_F=$F_reads;
$residue_R=$R_reads;
}
#print "then we sort it\n";
$count_a=0;
$count_c=0;
$count_g=0;
$count_t=0;
foreach (sort keys %hash){
$first=substr($_,0,1);
$len=length;
$last=substr($_,$len-1,1);
#print "$first\t$last\t$hash{$_}\n";
$count_a++ if $last eq 'A';
$count_c++ if $last eq 'C';
$count_g++ if $last eq 'G';
$count_t++ if $last eq 'T';
print "$last\t$hash{$_}\t$count_a\t$count_c\t$count_g\t$count_t\n";
}
unlink("tmp_forward.txt");
unlink("tmp_reverse.txt");
[/perl]
一、下载及安装软件
这个软件需要edu邮箱注册才能下载,可能是仅对科研高校开放吧。所以软件地址我就不列了。
它其实是几个perl程序,比较重要的是这个人类的数据库,snp注释必须的。
参考:http://annovar.readthedocs.org/en/latest/misc/accessory/
二,准备数据
既然是注释,那当然要有数据库啦!数据库倒是有下载地址
http://www.openbioinformatics.org/annovar/download/hg19_ALL.sites.2010_11.txt.gz
也可以用命令来下载
Perl ./annotate_variation.pl -downdb -buildver hg19 -webfrom annovar refGene humandb/
然后我们是对snp-calling流程跑出来的VCF文件进行注释,所以必须要有自己的VCF文件,VCF格式详解见本博客另一篇文章,或者搜索也行
http://vcftools.sourceforge.net/man_latest.html
三、运行的命令
首先把vcf格式文件,转换成空格分隔格式文件,自己写脚本也很好弄
perl convert2annovar.pl -format vcf
/home/jmzeng/raw-reads/whole-exon/snp-calling/tmp1.vcf >annovar.input
变成了空格分隔的文件
然后把转换好的数据进行注释即可
./annotate_variation.pl -out ex1 -build hg19 example/ex1.avinput humandb/
四,输出文件解读
需要插件和自己修改主题下面的foot.php代码。
参考 http://jingyan.baidu.com/article/ae97a646ce37c2bbfd461d01.html
步骤如下:
1、登陆到wp后台,鼠标移动到左侧菜单的“插件”链接上,会弹出子菜单,点击子菜单的“安装插件”链接
2、WP-PostViews插件显示wordpress文章点击浏览量
在“安装插件”链接页面的搜索框中输入“WP-PostViews”,然后回车
3、WP-PostViews插件显示wordpress文章点击浏览量
在搜索结果页面点击“WP-PostViews”插件内容区域的“现在安装”按钮
4、WP-PostViews插件显示wordpress文章点击浏览量
程序自动下载插件到服务器并解压安装,一直等到安装成功信息出现,然后在安装成功提示页面点击“启动插件”链接。
5、WP-PostViews插件显示wordpress文章点击浏览量
页面会自动跳转到“已安装插件”页面,在已安装插件列表中我们可以看到“Form Manager”插件已经处于启用状态(插件名下是“停用”链接)。
有了这个插件之后,我们的整个网页环境里面就多了一个 the_views()函数,它统计着每个文章的点击数,这样我们之前的网页就能显示点击数了。
这个是我现在用的主题的php代码,把文章用span标记隔开了,而且显示着上面php代码里面的每一个内容包括日期,分类,标签,评论等等
其中thez-view()这个函数返回的不仅仅是一个访客数,但是我的文章的访客都太少了,所以我写了一个脚本帮我刷一刷流量。
[perl]
use List::MoreUtils qw(uniq);
$page='http://www.bio-info-trainee.com/?paged=';
foreach (1..5){ #我的文章比较少,就42个,所以只有5个页面
$url_page=$page.$_;
$tmp=`curl $url_page`;
#@p=$tmp=~/p=(\d+)/;
$tmp =~ s/(p=\d+)/push @p, $1/eg; #寻找p=数字这样的标签组合成新的网页地址
}
@p=uniq @p;
print "$_\n" foreach @p; #可以找到所有42个网页的地址
foreach (@p){
$new_url='http://www.bio-info-trainee.com/?'.$_;
`curl $new_url` foreach (1..100); #每个网页刷一百次
}
[/perl]
大家可以看到这个网页被刷的过程,从15到21到27直到100
大家现在再去看我的网页,就每个文章都有一百的访问量啦!
http://www.bio-info-trainee.com/
下载地址我就不贴了,随便谷歌一下即可!
Genome Reference Consortium Human ---》 GRCh3
Feb. 2009 (hg19, GRCh37)这个是重点
Mar 2006 assembly = hg18 = NCBI36.
May 2004 assembly = hg17 = NCBI35.
July 2003 assembly = hg16 = NCBI34
以前的老版本就不用看啦,现在其实都已经有hg38出来啦,GRCh38 (NCBI) and hg38(UCSC)
参考:http://age.wang.blog.163.com/blog/static/119252448201092284725460/
http://www.ncbi.nlm.nih.gov/projects/genome/assembly/grc/human/
人的hg19基因组是3G的大小,因为一个英文字符是一个字节,所以也是30亿bp的碱基。
包括22条常染色体和X,Y性染色体及M线粒体染色体。
查看该文件可以看到,里面有很多的N,这是基因组里面未知的序列,用N占位,但是觉得部分都是A.T.C.G这样的字符,大小写都有,分别代表不同的意思。
然后我用linux的命令统计了一下里面这个文件的行数,
perl -lne 'END { print $. }' hg19.fa
awk 'END { print NR }' hg19.fa
wc -l hg19.fa
然后我写了一个脚本统计每条染色体的长度,42秒钟完成任务!
看来这个服务器的性能还是蛮强大的,读取文件非常快!
[perl]
while(<>){
chomp;
if (/>/){
if (exists $hash_chr{$key} ){
$len = length $hash_chr{$key};
print "$key => $len\n";
}
undef %hash_chr;
$key=$_;
}
else {
$hash_chr{$key}.=$_;
}
}
[/perl]
然后我用seed统计了一下hg19的词频(我不知道生物信息学里面的专业描述词语是什么)
我的程序耗费了42分钟才跑完,感觉我写的程序应该是没有问题的,让我吃惊的是总共竟然只有105万条独特的10bp短序列。然后我算了一下4的10次方,(⊙o⊙)…悲剧,原来只有1048576,之所以出现这种情况,是因为里面有N这个字符串,不仅仅是A.T.C.G四个字符。我用grep -v N seed10.txt |wc -l命令再次统计了一下,发现居然就是1048576,也就是说,任意A.T.C.G四个字符组成的10bp字符串短序列在人的基因组里面都可以找到!!!
然后我测试了一下,还是真是这样的,真是一个蛮有意思的现象。虽然我无法解释为什么,但是根据这个结果我们可以得知连续的A或者T在人类基因组里面高频出现,而连续的G或者C却很少!
如果我们储存这个10bp字符串的同时,也储存着它们在基因组的位置,那么就可以根据这个seed来进行比对,这就是blast的原理之一!
以前的一下perl代码分享
今天去参加了开源中国的一个源创会,感觉好隆重的样子,近五百人,BAT的工程师都过来演讲了,可都是数据库相关的, 我一个的都没有听懂,但是茶歇的披萨我倒是吃了不少。
说到开源中国,我想起来了我以前在上面分享的代码,上去看了看,竟然有那么多的访问量了,让我蛮意外的,那些代码完全是我学习perl的历程的真实写照。
首先,什么是BWT,可以参考博客
http://www.cnblogs.com/xudong-bupt/p/3763814.html
他讲的非常好。
一个长度为n的串A1A2A3...An经过旋转可以得到
A1A2A3...An
A2A3...AnA1
A3...AnA1A2
...
AnA1A2A3...
n个串,每个字符串的长度都是n。
对这些字符串进行排序,这样它们之前的顺序就被打乱了,打乱的那个顺序就是index,需要输出。
首先我们测试一个简单的字符串acaacg$,总共六个字符,加上一个$符号,下次再讲$符号的意义。
实现以上功能是比较简单的,代码如下

但是这是对于6个字符串等小片段字符串,如果是是几千万个字符的字符串,这样转换就会输出千万的平方个字符串组成的正方形数组,是很恐怖的数据量。所以在转换的同时就不能把整个千万字符储存在内存里面。
在生物学领域,是这样的,这千万个 千万个碱基的方阵,我们取每个字符串的前20个字符串就足以对它们进行排序,当然这只是近视的,我后面会讲精确排序,而且绕过内存的方法。
Perl程序如下
[perl]
while (<>){
next if />/;
chomp;
$a.=$_;
}
$a.='$';
$len=length $a;
$i=0;
print "first we transform it !!!\n";
foreach (0..$len-1){
$up=substr($a,0,$_);
$down=substr($a,$_);
#print "$down$up\n";
#$hash{"$down$up"}=$i;
$key=substr("$down$up",0,20);
$key=$key.”\t”.substr("$down$up",$len-1);
$hash{$key}=$i;
$i++;
}
print "then we sort it\n";
foreach (sort keys %hash){
$first=substr($_,0,1);
$len=length;
$last=substr($_,$len-1,1);
#print "$first\t$last\t$hash{$_}\n";
print "$_\t$hash{$_}\n";
}
[/perl]
运行的结果如下
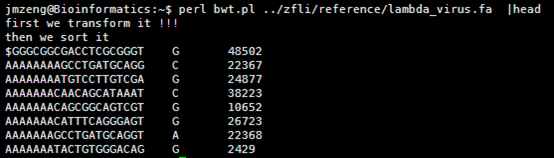
个人觉得这样排序是极好的,但是暂时还没想到如何解决不够精确的问题!!!
参考:
http://tieba.baidu.com/p/1504205984
http://www.cnblogs.com/xudong-bupt/p/3763814.html
在perl里面字符串跟数字是不区分的,所以写代码需要考虑到它们的区别!
@a=1..1000;
$b=56; #just a example
sub half_search{
my($ref,$key,$low,$high)=@_;
$high=@{$ref}-1 unless $high;
if ($key < $ref->[$low] or $key > $ref->[$high]){
print "not exists !!!\n";
last;
}
if ($ref->[$low] > $ref->[$high]){
print "not sort array !!!\n" ;
last;
}
$mid=int (($low+$high)/2);
if ($ref->[$mid] == $key) {
return $mid+1;
}
elsif ($ref->[$mid] < $key) {
&half_search($ref,$key,$mid+1,$high);
}
else{
&half_search($ref,$key,$low,$mid-1);
}
}
print &half_search(\@a,$b);
对排序好的数字数组来说,是非常简单的,非常快速。
@a=qw(a b c d e f g h );
$b='d';
sub half_search{
my($ref,$key,$low,$high)=@_;
$high=@{$ref}-1 unless $high;
if ($key lt $ref->[$low] or $key gt $ref->[$high]){
print "not exists !!!\n";
last;
}
if ($ref->[$low] gt $ref->[$high]){
print "not sort array !!!\n" ;
last;
}
$mid=int (($low+$high)/2);
if ($ref->[$mid] eq $key) {
return $mid+1;
}
elsif ($ref->[$mid] gt $key) {
&half_search($ref,$key,$mid+1,$high);
}
else{
&half_search($ref,$key,$low,$mid-1);
}
}
print &half_search(\@a,$b);
本人亲测可用,就不贴图啦!
生信常用论坛bio-star里面所有帖子爬取
这个是爬虫专题第一集,主要讲如何分析bio-star这个网站并爬去所有的帖子列表,及标签列表等等,前提是读者必须掌握perl,然后学习perl的LWP模块,可以考虑打印那本书读读,挺有用的!
http://seqanswers.com/ 这个是首页
http://seqanswers.com/forums/forumdisplay.php?f=18 这个共570个页面需要爬取
http://seqanswers.com/forums/forumdisplay.php?f=18&order=desc&page=1
http://seqanswers.com/forums/forumdisplay.php?f=18&order=desc&page=570
<tbody id="threadbits_forum_18">这个里面包围这很多<tr>对,
前五个<tr>对可以跳过,里面的内容不需要
仿写fastqc软件的部分功能(上)
前面我们介绍了fastqc这个软件的使用方法 http://www.bio-info-trainee.com/?p=95 ,这是一个java软件,但是有些人服务器没有配置好这个java环境,导致无法使用,这里我贴出几个perl代码,也能实现fastqc的部分功能
统一测试文件是illumina的phred33格式的fastq文件,共100000/4=25000条reads,读长都是101个碱基
程序名-fastq2quality.pl
使用命令:perl fastq2quality.pl SRR504517_1.fastq >quality.txt
功能: 把fastq格式的每条原始reads的第四行ascii码质量值,转换为Q值并输出一个矩阵,有多少条reads就有多少行,每条reads的碱基数就是列数。