文章:A Targetable EGFR-Dependent Tumor-Initiating Program in Breast Cancer , 因为bulk测序无法解决问题,所以作者选择了单细胞转录组测序策略:
Category Archives: cancer
10X genomics单细胞数据集探索
其官网公布了非常多的数据集: https://support.10xgenomics.com/single-cell-gene-expression/datasets 只需要简单填写邮箱即可下载,如下:
专门分析10x genomic公司的单细胞转录组数据的软件套件
10x Genomics方法巧妙使用了Barcoding(条形码)和Microfluidics(微流体)技术,在单细胞分离、扩增原理上具有明显的优势,能帮助研究人员实现对细胞群体的划分与细胞群体间基因表达差异的检测,是肿瘤细胞异质性、免疫细胞群体检测以及胚胎发育研究的黄金方法。
空间单细胞DNA测序
空间单细胞DNA测序技术:Topographic Single Cell Sequencing 发表于2018年CELL杂志,文章题目是:Multiclonal Invasion in Breast Tumors Identified by Topographic Single Cell Sequencing31449-6) 非常值得精读。
TET2突变是如何引起超甲基化
TET2突变是如何引起超甲基化
癌症病人体内会检测到不正常的甲基化现象。
TET2可以氧化5mC成为5hmC,进而通过其它机制形成5fC和5caC。
很多血液肿瘤病人的TET2基因突变了,同时会显示出全局的5hmC水平下降。
有趣的是,全局的5hmC水平下降同样发生在很多实体肿瘤病人身上,但是那些病人很少有TET2突变发生。
那么,TET2突变,或者全局的5hmC水平下降,是如何导致启动子区域的CG岛的甲基化水平上升的呢?
有其它文献报道 hypermethylation和oxidative stress (OS)有关系
作者认为 oxidative stress (OS) 在其中起了关键的作用。
外显子测序流程-文章里面的
就是做一个图床而已,需要这个图片的网页url链接,没别的意思! Continue reading
it’s extremly easy to use firehose_get to download TCGA data
it's extremly easy to use firehose_get to download TCGA data
About firehose
This website is maintained by Broad Institute, the url is : http://firebrowse.org/ , and all of the TCGA data are stored in below urls : Continue reading
单个样本如何做拷贝数变异分析
ESCC-肿瘤空间异质性探究
ESCC-肿瘤空间异质性探究
肿瘤异质性包括空间异质性、时间异质性、解剖异质性、结构异质性、基因异质性和功能异质性等等
肿瘤异质性是恶性肿瘤的特征之一,是指肿瘤在生长过程中,经过多次分裂增殖,其子细胞呈现出分子生物学或基因方面的改变,从而使肿瘤的生长速度、侵袭能力、对药物的敏感性、预后等各方面产生差异。肿瘤异质性一直是肿瘤治疗的挑战之一,肿瘤内部不同亚群的细胞对药物敏感性的不同可能会导致治疗的失败。现在主流的探究肿瘤异质性的方法是:
- 对肿瘤病人的肿瘤组织进行不同时间点取样
- 对肿瘤病人的肿瘤组织不同部分分别取样
肿瘤全外显子测序数据分析流程大放送
这个一个肿瘤外显子项目的文章发表并且公布的公共数据,我这里给出全套分析流程代码。只需要你肯实践,就可以运行成功。
PS:有些后起之秀自己运营公众号或者博客喜欢批评我们这些老人,一味的堆砌代码不给解释,恶意揣测我们是因为不懂代码的原理。我表示很无语,我写了3千多篇教程,如果一篇篇都重复提到基础知识,我真的做不到。比如下面的流程,包括软件的用法,软件安装,注释数据库的下载,我博客都说过好几次了,直播我的基因组系列也详细解读过,我告诉你去哪里学,你却不珍惜,不当回事,呵呵。
癌症基因的somatic mutation calling 流程的评价体系
癌症基因的somatic mutation calling 流程的评价体系
文章是:A comprehensive assessment of somatic mutation detection in cancer using whole-genome sequencing
TCGA CNV全攻略
TCGA CNV全攻略
明白什么是CNV
对正常人来说,基因组应该是二倍体的,所以凡是测到非2倍体的地方都是CNV。但是CNV本身就是人群遗传物质多样性的体现,所以对癌症样本来说,是需要过滤掉正常人体内的germline的CNV,得到somatic的CNV。
下载TCGA所有癌症的maf文件做signature分析
下载TCGA所有癌症的maf文件做signature分析
TCGA计划的4个找somatic mutation的软件使用体验
TCGA计划的4个找somatic mutation的软件使用体验
体细胞突变(somatic mutation)是指患者某些组织或者器官后天性地发生了体细胞变异,虽然它不会遗传给后代个体,却可以通过细胞分裂,遗传给子代细胞。体细胞突变对肿瘤的发生发展有关键性的作用,并且它也是制定肿瘤癌症靶向治疗措施的关键所在。NGS使体细胞变异的检测更加全面,成本更低,在检测多种体细胞变异上具有很大的优势,但在使用过程中还存在着挑战:如样品降解、覆盖度不足、遗传异质性和组织污染(杂质)等问题。 为应对以上挑战,降低错误率,科学家采取了不同的算法和统计模型用于检测体细胞突变。目前最受欢迎的有Varscan、SomaticSniper、 Strelka 和MuTect2 。
下载TCGA所有癌症的maf文件计算TMB
下载TCGA所有癌症的maf文件计算TMB
明白什么是TMB
TMB (Tumor mutation burden)的定义:癌症样本全基因组中去除胚系DNA变异后体细胞突变数目。比如Lawrence,MS团队在Nature上发表的研究中,将超过100个突变/Mb称之为高TMB。 Continue reading
一个标准的TCGA大文章应该做哪些数据?
TCGA表达数据的多项应用之4–求指定基因在指定癌症里面的表达量相关性矩阵,与所有的基因比较。
这个不出图,会给出TCGA里面涉及到的所有基因跟你指定的基因的表达量相关系数和P值,分别你一次性的看清楚你感兴趣的基因跟体内其它基因在该癌症种类的相关性,当然,相关非因果,请谨慎应用! Continue reading
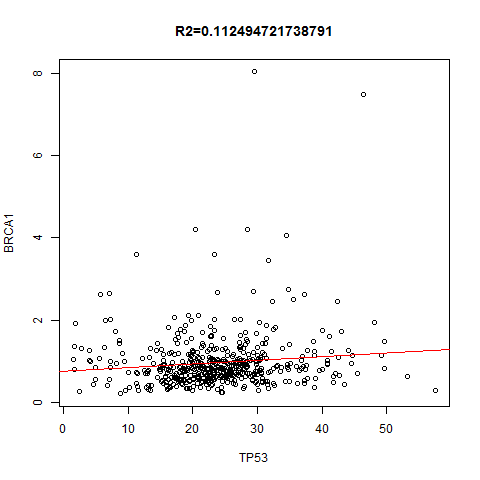
TCGA表达数据的多项应用之3–对指定的两个基因,在所有癌种里面找到correlation并作图
上面是指定一个基因在不同的癌种里面,本次讲指定任意两个基因,在所有癌种里面找到correlation并作图!图如下:
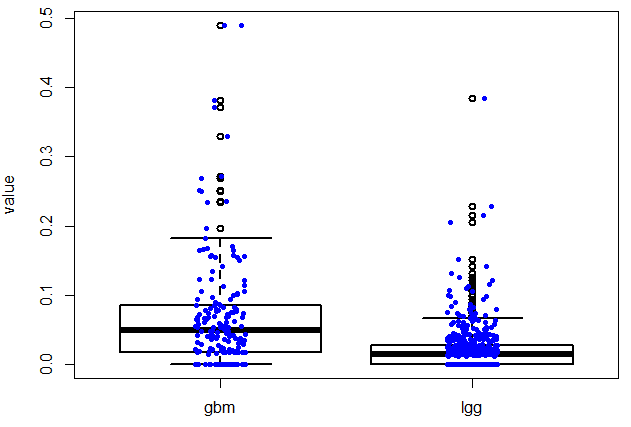
TCGA表达数据的多项应用之2–对指定基因在不同癌种里面画boxplot,或者在所有的normal组织里面看表达量!
好像文章题目没有长度限制,太好了!本讲所实现的目标非常简单,如题,指定基因在不同癌种里面画boxplot,或者在所有的normal组织里面看表达量!下面是一个具体的例子: