我在生信技能树多次分享过生存分析的细节;
Category Archives: 未分类
T细胞可以简单分成4类
最近在《生信技能树》公众号后台接到了求助,说他的肿瘤单细胞划分亚群的时候对T细胞的细分亚群把握不好,有超级简单的,也有非常复杂的,对于这样的求助,我也爱莫能助啊! Continue reading
TCGA各大癌症的somatic突变全景图鸟瞰
前面我们开通了明码标价专栏:
Y叔承包了富集分析结果的可视化
不管是 GO或者KEGG这样的常见数据库的注释结果,还是mesh,reactomePA, DOSE这样的略微小众的数据库资源,不管是超几何分布检验的富集还是GSEA的算法,都Y叔都有对应的可视化函数支持。
看肿瘤转移机制就比较N0和N3时期样品
众所周知,肿瘤的TNM分期是目前临床上比较常用的分期方式之一:
Continue reading
可视化单细胞亚群的标记基因的5个方法
好的颜值,人人都爱,是你接触有趣的灵魂的敲门砖。单细胞数据分析也是如此,人人都知道需要降维聚类分群。 Continue reading
流式分选后指定细胞亚群的表达量差异(无需单细胞)
传统的bulk转录组测序并没有没落,虽然说大家都在抢单细胞的热点!
没有差异分析也要做富集分析
最近看到了一个胰腺癌的单细胞文章,公开了其测序数据及表达量矩阵,很方便做图表复现。 Continue reading
RNA-Seq数据的内含子保留分析
前面我们的明码标价之普通转录组上游分析,受到了各大热心粉丝的吐槽,觉得太简单了我们居然还好意思收费。后面我就就加上了稍微有一点难度的《可变剪切》,不过仍然是阻挡不了粉丝无穷无尽的需求,后台有人发给我一个RNA-Seq数据的内含子保留分析需求。
我看了看粉丝发过来的文章,发表于 January 2021, 在CELL杂志的文章《Spliceosome-targeted therapies trigger an antiviral immune response in triple-negative breast cancer》,链接是:https://doi.org/10.1016/j.cell.2020.12.031
这个文章数据比较多:
SUM159 SD6 RNA-Seq #GSE163414
LM2 SD6 RNA-Seq #GSE163411
SUM159 Cytoplasmic RNA-Seq #GSE163232
SUM159 J2 dsRIPseq #GSE163188
Syngeneic model RNA-Seq #GSE163181
可以看到,主要是RNA-Seq数据啦,有两个是普通的细胞系处理前后的表达量差异情况探索,所以出图如下:
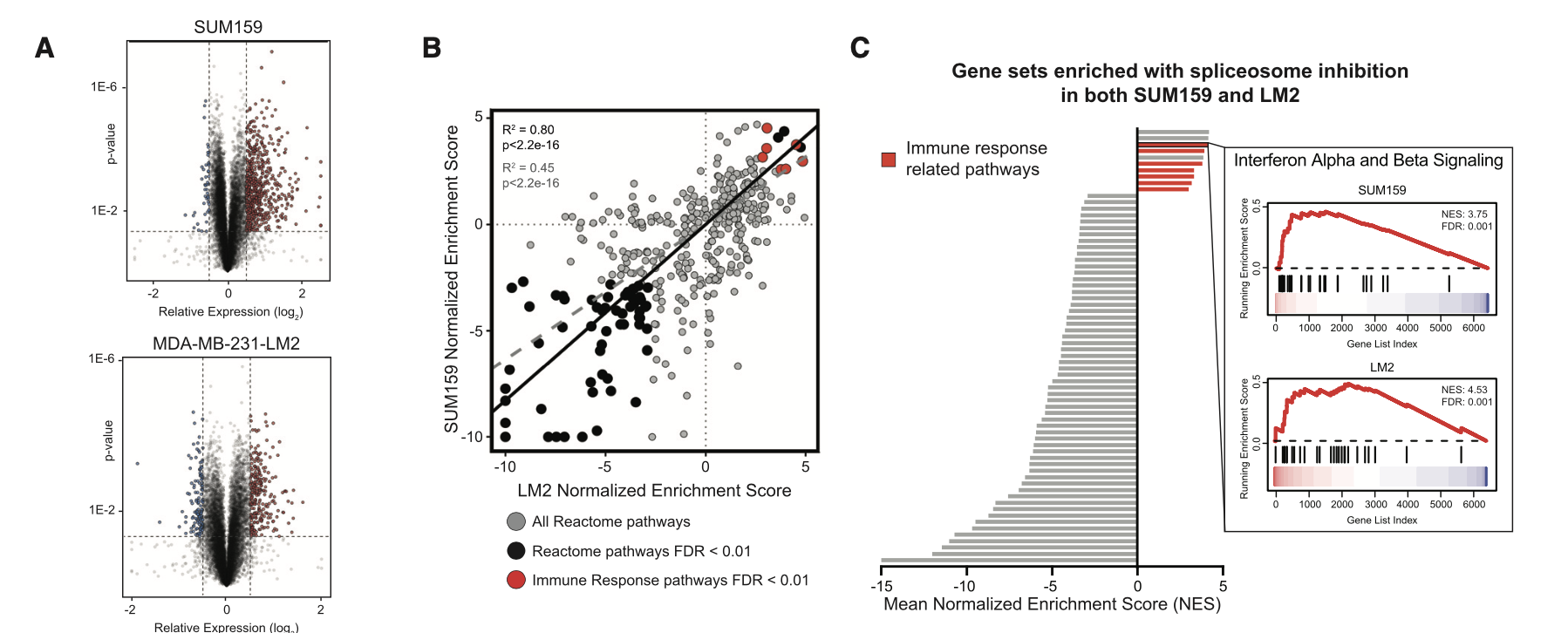
这个已经是超级简单了, 我们的明码标价之转录组常规测序服务(仅需799每个样品) 和 明码标价之普通转录组上游分析 就是对这样的 RNA-Seq拿到了表达量矩阵,然后下游分析也是平淡无奇,仅收费800,代码呢,我也多次分享了,基本上看我六年前的表达芯片的公共数据库挖掘系列推文即可;
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
这样的分析流程基本上绝大部分粉丝已经是无需委托我们啦,所以粉丝发给我的是 RNA-Seq数据的内含子保留分析需求,步骤如下:
- Hisat2-aligned reads were filtered for proper-paired reads (-f 2 flag in SAMtools).
- Intron annotations were parsed from UCSC RefSeq gene annotation files and were filtered to exclude features that overlap genomic loci on the same strand.
- Reads mapping to introns were counted using Pysam.
- For each intron feature, we defined the following two read classes:
- (1) ‘‘intronic’’ reads mapping at least 6 bases contiguously within the intron
- (2) ‘‘spanning’’ reads with ends mapping to the flanking exons.
- The intron retention (IR) score was then computed as the ratio of the RPKM-normalized ‘‘intronic’’ read density over the RPKM-normalized ‘‘spanning’’ read den- sity.
- In order to compare commonly expressed IR events across samples, introns with < 10 spanning RPKM in any sample were excluded from all analyses.
对我们有ngs组学数据分析经验的人来说,其实并不难,无非就是安装几个软件,使用几个包。但对于没有学过编程的纯生物学研究者来说基本上不可能完成,也没有这样的网页工具。
但是呢,这个流程又确实是过于个性化,哪怕对我们来说很简单,也其实是耗费时间和精力需要研发调试的。
首先你需要有RNA-seq的fastq文件
如果是TCGA数据库,步骤如下:
- Intron retention analysis was performed on BRCA TCGA RNA sequencing datasets (Koboldt et al., 2012).
- TCGA fastq reads were mapped using the STAR aligner (v2.3.1) (Dobin et al., 2013) onto the hg19/GRCh37 reference genome as previously described (Hsu et al., 2015).
- Level of intron retention (IR level) within each sample was calculated as the number of introns with IR scores > 0.01, as defined previously.
- ‘‘High’’ and ‘‘Low’’ IR were defined as having an IR level outside one standard deviation of the mean.
- RSEM normalized gene expression data from TCGA was obtained from the Broad GDAC Firehose.
一般来说,大家是很难下载TCGA数据库原始fastq文件,这个权限审核比较严厉,不过咱们数据挖掘呢完全没有毕业一直盯着TCGA数据库啊,自己领域的普通RNA-seq肯定也是不少。如果是认真搞科研,你一定会自行调研和阅读文献,找到合适的数据集。
数据挖掘的核心就是通过分类把基因数量搞少
部分粉丝看到这里,可能无法理解RNA-Seq数据的内含子保留分析的意义是什么?
其实就是多了一个维度的指标,来把你的样本分类,分类后就可以找差异。同样的我们可以看这个示例文章,感觉每个样品的IR指标,把病人分成IR高低两个组别,然后走普通的ssGSEA分析,生存分析。
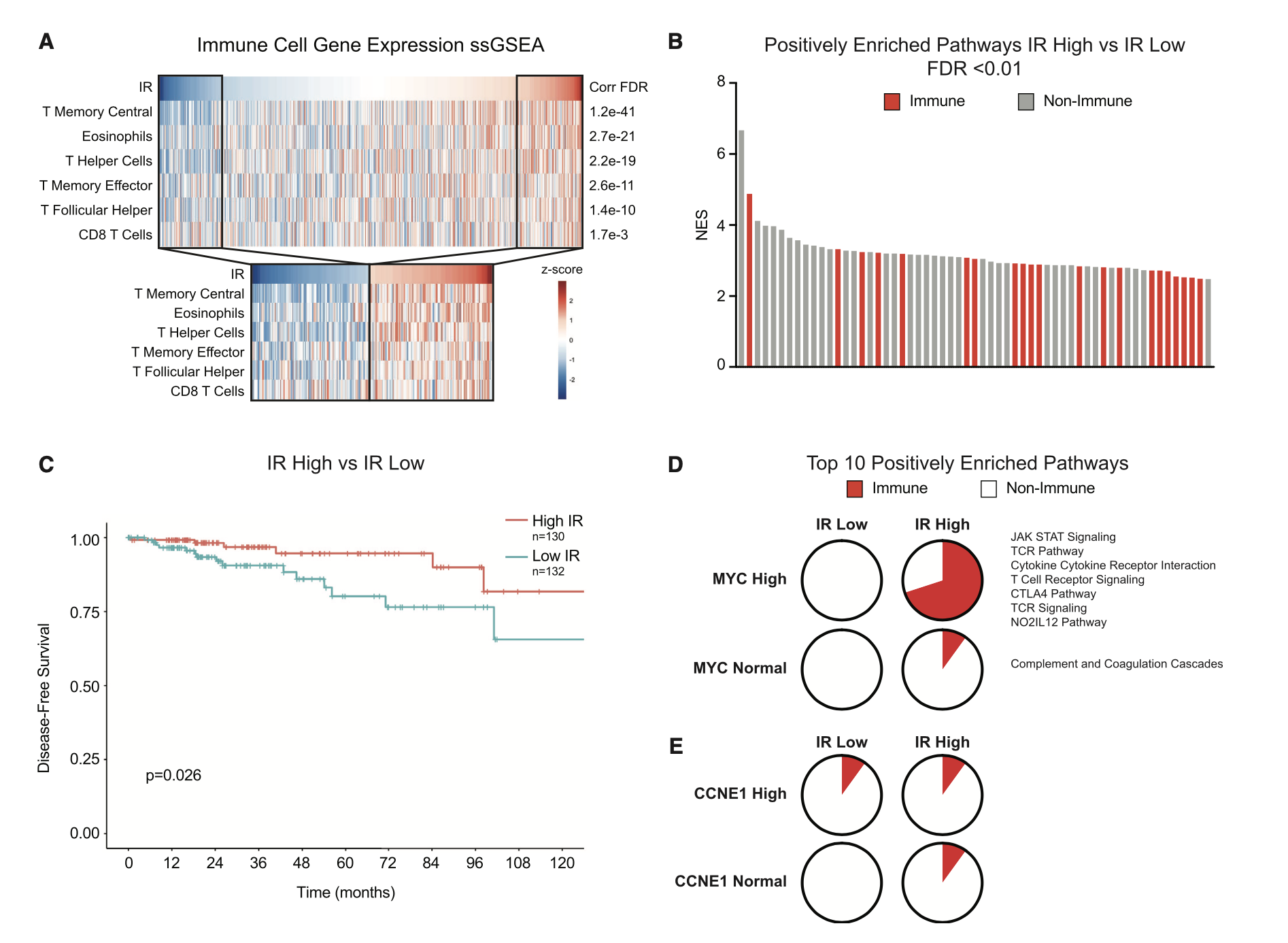
这一套组合拳,大家是不是很眼熟啊?
如果你也想做自己的的RNA-Seq数据的内含子保留分析,赶快联系我们吧,同样的,我们的分析仍然是明码标价,单个RNA-Seq数据的内含子保留分析收费仅需800元,因为是纯粹的基于Linux平台的各种软件脚本,所以提供你全套数据和脚本但是无法保证你能运行成功,因为你不一定有自己的服务器。
你的科研也是在划水吗
最近看到了朋友圈疯转的:[发paper怎么少走弯路?顶刊编辑手把手指点!【文字版】] Continue reading
去broad官网下载msigdb数据库文件很麻烦
我在:借鉴escape包的一些可视化GSVA或者ssGSEA结果矩阵的方法 和 对单细胞表达矩阵做gsea分析的两个教程里面提到过,MSigDB(Molecular Signatures Database)数据库中定义了已知的基因集合:http://software.broadinstitute.org/gsea/msigdb 需要注册才能下载。
但是这个GitHub包,ncborcherding/escape文档,在:http://www.bioconductor.org/packages/release/bioc/vignettes/escape/inst/doc/vignette.html 提供了一个封装好的MSigDB数据库信息,其实你仔细看它的文档,它的打包其实是依赖于msigdbr_7.2.1。
获取 MigDB中的全部基因集
MigDB中的全部基因集 都被这个GitHub包,ncborcherding/escape 打包起来了,MSigDB(Molecular Signatures Database)数据库中定义了已知的基因集合:http://software.broadinstitute.org/gsea/msigdb 包括H和C1-C7八个系列(Collection),每个系列分别是:
- H: hallmark gene sets (癌症)特征基因集合,共50组,最常用;
- C1: positional gene sets 位置基因集合,根据染色体位置,共326个,用的很少;
- C2: curated gene sets:(专家)校验基因集合,基于通路、文献等:
- C3: motif gene sets:模式基因集合,主要包括microRNA和转录因子靶基因两部分
- C4: computational gene sets:计算基因集合,通过挖掘癌症相关芯片数据定义的基因集合;
- C5: GO gene sets:Gene Ontology 基因本体论,包括BP(生物学过程biological process,细胞原件cellular component和分子功能molecular function三部分)
- C6: oncogenic signatures:癌症特征基因集合,大部分来源于NCBI GEO 发表芯片数据
- C7: immunologic signatures: 免疫相关基因集合。
GS <- getGeneSets(library = "H")
GS
MigDB中的全部基因集 被 构建成为: a list of GSEABase GeneSet objects ,获取 hallmark gene sets (癌症)特征基因集合。
源头是msigdbr 包
安装方法非常简单:
install.packages("msigdbr")
但是这个msigdbr并没有我想象中的那么大:
Installing package into ‘C:/Users/win10/Documents/R/win-library/4.0’
(as ‘lib’ is unspecified)
试开URL’https://cran.rstudio.com/bin/windows/contrib/4.0/msigdbr_7.2.1.zip'
Content type 'application/zip' length 6737651 bytes (6.4 MB)
downloaded 6.4 MB
package ‘msigdbr’ successfully unpacked and MD5 sums checked
同样的,学习R包,看看文档即可,在: https://cran.r-project.org/web/packages/msigdbr/vignettes/msigdbr-intro.html
Documentation for package ‘msigdbr’ version 7.2.1
DESCRIPTION file.
User guides, package vignettes and other documentation.
Help Pages
msigdbr Retrieve the gene sets data frame
msigdbr_collections List the collections available in the msigdbr package
msigdbr_show_species List the species available in the msigdbr package
msigdbr_species List the species available in the msigdbr package
这些代码使用就明白了,确实没啥好继续讲解的:
library(msigdbr)
# All gene sets in the database can be retrieved without specifying a collection/category.
all_gene_sets = msigdbr(species = "Mus musculus")
head(all_gene_sets)
msigdbr_species()
all_gene_sets = msigdbr(species = "Homo sapiens")
无非就是封装和对象,前面的 escape 包提供了getGeneSets函数,我们的这个msigdbr提供了 msigdbr函数。
生信基石之R语言
B站的10个小时教学视频务必看完,参考 GitHub 仓库存放的相关学习路线指导资料:https://github.com/jmzeng1314/R_bilibili ,可以参考一些优秀笔记,比如https://mubu.com/doc/2KUiSCfVsg
- 初级10 个题目:http://www.bio-info-trainee.com/3793.html
- 中级要求是:http://www.bio-info-trainee.com/3750.html
- 高级要求是完成20题: http://www.bio-info-trainee.com/3415.html
- 统计专题 30题:http://www.bio-info-trainee.com/4385.html
- 可视化专题30题:http://www.bio-info-trainee.com/4387.html
文末友情推荐
全套MeRIP-seq文章图表复现代码
前面我们分享了 跟着Nature Medicine学MeDIP-seq数据分析,数据和代码都是公开,这个2G的压缩包文件,足以学习3个月,写60篇教程。 Continue reading
人类蛋白质计划,希望有你!
我们《生信技能树》早期也分享过蛋白质组学数据处理教程,目录如下:
Continue reading
人类研究有GTEx数据库那么大鼠和小鼠研究呢
GTEx数据库想必大家并不陌生了,通常我们在挖掘TCGA数据库的时候,会发现该项目纳入的正常组织测序结果是非常少的,也就是说很多病人都不会有他的正常组织的转录组测序结果。 Continue reading
单细胞转录组数据分析流程的每一个步骤都值得一个综述
四年前我做了一个单细胞课程,就是对scRNAseq包里面的数据示例的一些处理。 Continue reading
单细胞转录组下游分析是否有必要删除线粒体和核糖体基因
如果你看了我的单细胞转录组数据分析的 基础10讲:
- 01. 上游分析流程
- 02.课题多少个样品,测序数据量如何
- 03. 过滤不合格细胞和基因(数据质控很重要)
- 04. 过滤线粒体核糖体基因
- 05. 去除细胞效应和基因效应
- 06.单细胞转录组数据的降维聚类分群
- 07.单细胞转录组数据处理之细胞亚群注释
- 08.把拿到的亚群进行更细致的分群
- 09.单细胞转录组数据处理之细胞亚群比例比较
会发现,里面虽然是根据线粒体和核糖体基因的比例,对细胞进行了过滤, 但是并没有改变基因的数量。基因的数量主要是在过滤那些在绝大部分细胞都不表达的,然后挑选高变基因,这样的降维操作。
但是,关于单细胞转录组下游分析是否有必要删除线粒体和核糖体基因讨论却经常性的出现在各大单细胞交流群,比如发表于2021年3月的文章,标题是:《Single-cell analyses of Crohn’s disease tissues reveal intestinal intraepithelial T cells heterogeneity and altered subset distributions》,链接是:https://www.nature.com/articles/s41467-021-22164-6 就做了这样的操作:

其单细胞表达量矩阵是公开可以获取的:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE157477
我们首先发起一个讨论,大家是否赞成在单细胞转录组下游的降维聚类分群之前进行删除线粒体和核糖体基因的操作。
另外,提出来一个学徒作业,参考前面的例子:人人都能学会的单细胞聚类分群注释 ,对 GSE157477的单细胞表达量矩阵进行两次单细胞转录组下游的降维聚类分群,首先是不进行删除线粒体和核糖体基因,然后是删除线粒体和核糖体基因后的表达量矩阵,对这两次的降维聚类分群结果进行对比!
Continue reading
当你的单细胞亚群都不特异的时候肿么办
做单细胞数据分析,我们当然希望看到一个清晰的降维聚类分群结果,这样才方便做生物学亚群注释,比如前面的例子:[人人都能学会的单细胞聚类分群注释] Continue reading
吊打ChIP-seq的CUT&Tag技术
看到了发表于2020年8月的一个研究,标题是《Efficient chromatin profiling of H3K4me3 modification in cotton using CUT&Tag》,链接是 https://link.springer.com/article/10.1186/s13007-020-00664-8 Continue reading
都不需要正式发布在bioconductor的包也可以发文章了
众所周知,发布在bioconductor的包主要是生物信息学相关,在官方可以看到其主要是分成3类: Continue reading
肺的正常上皮细胞可以分成这5群
绝大部分的肿瘤研究单细胞研究我介绍过 [CNS图表复现08—肿瘤单细胞数据第一次分群通用规则] Continue reading