还记得几年前前准备单细胞课程作业,查遍全网基本上找不到中文资料,甚至英文文献都少得可怜,虽然那个时候单细胞就已经显现出热点的趋势,大多数CNS之作,但是在癌症领域仅仅是6个癌症类型有单细胞转录组技术应用的研究,不像现在,大量的CNS犹如雨后春笋般冒出来。
这些年陆陆续续阅读了近百篇该领域的CNS文献,所以大概总结了单细胞转录组技术肿瘤研究3大应用方向 Continue reading
Category Archives: 未分类
单细胞可以跟bulk转录组结合起来
记得上半年参加某个单细胞会议,深圳某医院科研负责人(院长吧)豪气的说,大家尽管来我们医院,保底50万年薪,而且咱们这里单细胞平台全部搭建好了,以后只做单细胞,所有之前使用bulk转录组的实验统统换成单细胞。 Continue reading
单细胞基础课程练习题
单细胞基础视频课程结束了,不知道大家学的怎么样,看起来线下学徒和实习生都学的挺好的,还有详细的笔记分享,考虑到大部分人是没有机会线下接受指导,有必要出一份考题督促或者提醒大家赶紧学完基础课程!全网第一个单细胞课程(基础)满一千份销量就停止发售 Continue reading
单细胞不只是10X,还有CyTOF
最近看到文章里面出现了单细胞转录组测序的tSNE图,如下:
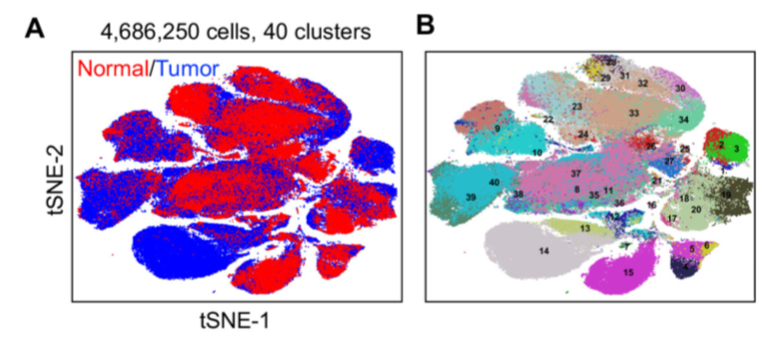
看到如此巨大的细胞数量,本来以为是10X,结果仔细看文章,作者利用CyTOF技术对HCC的免疫微环境异质性进行了分析,约四百万个免疫细胞呈现出40种聚类。 Continue reading
从一个**被更新后的GTF文件**得到geneID和gene类型的对应关系
上个月听了jimmy老师的巡讲后,开始学习GEO数据挖掘,看了相关视频后,想实战一把,于是看了jimmy老师去年开始分享的GEO数据挖掘帖子,没想到第一期就碰到个钉子,还好自己解决了,以下是分享。 Continue reading
猜一猜这个人类肺部器官单细胞研究会发在CNS吗
最近在bioRxiv 预印本发现了一篇人类肺部器官单细胞研究,来自于斯坦福大学团队,发表在Aug. 27, 2019; doi: http://dx.doi.org/10.1101/742320 有约7万个单细胞数据,找到了58个细胞亚群,其中大部分是前人研究发现过的,但是有14个新亚群。 Continue reading
不止是NCBI的SRA可以下载测序数据
大家看我在生信技能树发布的各个NGS组学的视频教程, 基本都是随手找到一篇文章,就去查询其原始数据,通常是在NCBI的SRA,然后使用prefetch下载sra文件,有的时候还好使用aspera进行加速。 Continue reading
不同转录组流程结果到底该如何比较
最近学徒刚刚完成了RNA-seq的训练,我就随机抽取了一个公共数据库项目给他作为作业,研究者是通过CRISPR/Cas9对 nasopharyngeal carcinoma (NPC) 细胞系的p53引入了一个杂合突变 R280T ,然后看mRNA profiles of CNE2 (control) and KO CNE2 cells 的差异。 Continue reading
不是转录组就应该有全局差异
转录组已经走入寻常百姓家了,现在生命科学领域实验设计搞个转录组测序就跟PCR和WB一样频繁和普及,但并不是所有人都会分析,我在全国巡讲分享生物信息学经验的时候特别指出了两个容易混淆的知识点,但大伙类似的错误认知还有不少,这里再详细讲解一个,也顺便diss一些已经发表的文章。 Continue reading
病理差异分析是否需要排除组织差异呢?
看到文章:Mol Biol Cell. 2004 Jun; 题目是:Different gene expression patterns in invasive lobular and ductal carcinomas of the breast. 使用基因芯片技术,测了 21 ILCs, 38 IDCs, two lymph node metastases, and three normal tissues 样本的表达信息,最后全文的落脚点是 ILCs 和 IDCs的差异表达情况: Continue reading
10X转录组数据是否可以inferCNV呢
有学员问10x的3‘端测序来infer CNV 是否可靠?
这个问题,说实话,很难回答,因为要是能完整回答这个问题,其实就是一篇正经的生物信息学文章了。 Continue reading
4年前的TCGA重磅资料你学了吗
还记得当初在微信公众号已经成为了一片红海的时候,我们生信技能树由论坛和博客这样的过时的互联网形态转型杀进来微信公众号自媒体战场,当时奋斗的非常辛苦,虽然现在已经成为生物信息学领域当之无愧的流量当担。 Continue reading
植物种子发育转录调控-2020年8月份第4周(总第128周 )
本次分析的文章发表在Plant Cell. 2019 May; 题为‘High-temporal-resolution Transcriptome Landscape of Early Maize Seed Development’的学术论文。该研究通过对玉米珠心(包括胚囊)进行RNA-seq分析,绘制了高时间分辨率的玉米籽粒发育早期转录组图谱。 需要注意的是 days after pollination (DAP) 和 hours after pollination (HAP) Continue reading
乳腺癌浸润T细胞的单细胞RNA测序 -(2019年8月份)第32周(总第80周 )
发表在 Nature Medicine (2018) ,标题是:Single-cell profiling of breast cancer T cells reveals a tissue-resident memory subset associated with improved prognosis 作者从3个乳腺癌患者体内通过FACS筛选到乳腺癌中肿瘤浸润淋巴细胞(TILs),使用商业仪器10X来做单细胞转录组,得到6,311个T细胞数据。 Continue reading
单细胞转录组对乳腺上皮细胞进行更细致的分类 -(2019年8月份)第31周(总第79周 )
文章发表于2018年的《Nature Communications》 , 题目为“Profiling human breast epithelial cells using single cell RNA sequencing identifies cell diversity”。 作者从7个个体的乳腺上皮细胞提取25,790 个单细胞进行转录组测序。测序的原始数据都上传到了NCBI的GEO数据库: GSE113197.如下; Continue reading
单细胞转录组探索小鼠乳腺发育 -(2019年8月份)第30周(总第78周 )
2017两篇文章陆续发表在nature communication杂志,都是关于小鼠乳腺发育的,所以一起解读
首先是发表于2017年9月的NC,文章是;Construction of developmental lineage relationships in the mouse mammary gland by single-cell RNA profiling 系统性的跟踪检测了小鼠epithelial cells 的各种时期的单细胞转录组 Continue reading
肺癌单细胞图谱 -(2019年8月份)第29周(总第77周 )
本次分享的文章发表在 Nat Med. 2018 Aug; 题目是:Phenotype molding of stromal cells in the lung tumor microenvironment. 共选取5例病人的共19个样本,通过10×genomics单细胞转录组测序探索基质细胞的亚群分类、基因功能(信号通路)、关键marker基因和临床预后。共鉴定出52个基质细胞亚群,反映了肿瘤微环境复杂性。对基质细胞的marker基因做生存曲线,发现这些marker基因可以作为肺癌预后诊断的潜在标志物。 为科学家们提供一种研究细胞类型的基准,便于后期研究人员在体外或动物模型中对这些细胞类型进行深度研究。同时,这些基质细胞亚型的生物标记作用,对患者的治疗与预后评估也至关重要。 Continue reading
乳腺癌微环境不同免疫表型单细胞分布 -(2019年7月份)第28周(总第76周 )
占位!
挑选METEBRIC和TCGA数据库的TNBC重新分类-(2019年7月份)第27周(总第75周 )
文章发表于 Annals of Oncology, April 2018, 链接是,https://doi.org/10.1093/annonc/mdy024
主要是下载两个数据库总共 (n = 550) 的TNBC病人的3数据进行分析,即:
- Molecular Taxonomy of Breast Cancer International Consortium (METABRIC)
- The Cancer Genome Atlas. TNBC samples
这3种数据分别是: - copy-number aberrations
- somatic mutations
- gene expression
进行了整合分析,所谓的多组学整合,其实就是根据表达数据对样本进行分组, 然后看CNV和SNV层面的差异。 Continue reading
6个TNBC病人单细胞转录组 -(2019年7月份)第26周(总第74周 )
发表于2018的NC,文章是:Unravelling subclonal heterogeneity and aggressive disease states in TNBC through single-cell RNA-seq 对6个TNBC病人总共测了 超过1500个单细胞 ,质控后还剩下1189个单细胞进入下游分析。使用的是FACS加上Smart-seq2 ,非常中规中矩的分析,所以就发了同样中规中矩的NC。 Continue reading