基于高通量测序数据进行HLA分型的软件挺多的,比较老的有三个,作者分别是Boegel et al.Kim et al.Major et al.,然后他们都被OptiType这个软件的作者被批评了,我这里先介绍Kim et al的seq2HLA使用方法,以下是它的一些链接。
功能概述
seq2HLA is a computational tool to determine Human Leukocyte Antigen (HLA) directly from existing and future short RNA-Seq reads. It takes standard RNA-Seq sequence reads in fastq format as input, uses a bowtie index comprising known HLA alleles and outputs the most likely HLA class I and class II types, a p-value for each call, and the expression of each class.
软件简介
Type of tool Program
Nature of tool Standalone
Operating system Unix/Linux, Mac OS X
Language Python, R
Article (Boegel et al., 2013) HLA typing from RNA-Seq sequence reads. Genome medicine.
PubMed http://www.ncbi.nlm.nih.gov/pubmed/23259685
URL https://bitbucket.org/sebastian_boegel/seq2hla
源代码,下载并安装
https://bitbucket.org/sebastian_boegel/seq2hla/src
http://tron-mainz.de/tron-facilities/computational-medicine/seq2hla/
第一版是这样的
第二版是这样的
只有第二版才支持gz压缩包格式的fastq,而且不需要指定length了
其中reference文件夹下面的是发布这个软件的团体已经制备好来的HLA库文件
下载即可使用,前提是你的系统其它环境都OK
用法:
python seq2HLA.py -1 <readfile1> -2 <readfile2> -r "<runname>" [-p <int>]* [-3 <int>]**
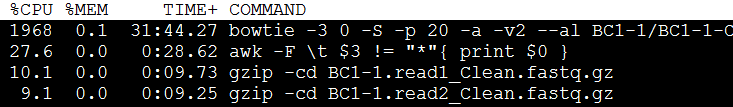
很简单,-1和-2指定我们的双端测序数据即可,可以是压缩包格式的(自动调用gzip),-r的输出目录,会输出7个文件,需要一个个解读,-p指定线程数给bowtie用的,-3是指定需要trim几个低质量碱基。
但是运行这个软件的要求非常多,需要安装好python和R,而且还有版本限制,需要安装好biopython而且还必须是双端测序,而且当前文件夹下面的reference文件夹下面必须有参考基因组的bowtie索引,而且系统必须安装好了bowtie,还需要在快捷方式里面!
我这里用的是第二版的
所以,我用的也是第二版改进的命令。非常好用,我这里用的是一个外显子测序数据,是hiseq2500测的PE100
python seq2HLA.py -1 ../../6-exon/PC3-1.read1_Clean.fastq.gz -2 ../../6-exon/PC3-1.read2_Clean.fastq.gz -r PC3
貌似输出文件太多了一点
#Output:#The results are output to stdout and to textfiles. Most important are:
#i) <prefix>-ClassI.HLAgenotype2digits => 2 digit result of Class I
#ii) <prefix>-ClassII.HLAgenotype2digits => 2 digit result of Class II
#iii) <prefix>-ClassI.HLAgenotype4digits => 4 digit result of Class I
#iv) <prefix>-ClassII.HLAgenotype4digits => 4 digit result of Class II
#v) <prefix>.ambiguity => reports typing ambuigities (more than one solution for an allele possible)
#vi) <prefix>-ClassI.expression => expression of Class I alleles
#vii) <prefix>-ClassII.expression => expression of Class II alleles
根据文献,我简单看了一下,文件的确好复杂,不过我们只需要看输出日志即可
-----------2 digit typing results-------------
#Locus Allele 1 Confidence Allele 2 Confidence
A A*68 7.287148e-05 A*24 0.03680272
B B*52 0.1717737 B*53 0.3952319
C C*12 0.03009331 hoz("C*14") 0.6783964
Calculation of locus-specific expression ...
BC1-1/BC1-1-ClassI.bowtielog
A: 7.93 RPKM
C: 9.75 RPKM
B: 8.35 RPKM
The digital haplotype is written into BC1-1/BC1-1-ClassI.digitalhaplotype3
-----------4 digit typing results-------------
#Locus Allele 1 Confidence Allele 2 Confidence
!A A*68:01 7.287148e-05 A*24:02 0.03680272
!B B*52:01 0.1717737 B*53:01' 0.6542288
!C C*12:02 0.03371717 C*12:02 0.6783964
上面的HLA的class I的数据结果
接下来是class II的数据结果,是不是很简单呀!
-----------2 digit typing results-------------
#Locus Allele 1 Confidence Allele 2 Confidence
DQA DQA1*01 0.1511134 DQA1*02 0
DQB DQB1*02 0.02321615 DQB1*05 0.42202
DRB DRB1*15 2.595144e-05 DRB1*07 0.321219
Calculation of locus-specific expression ...
BC1-1/BC1-1-ClassII.bowtielog
DQB1: 4.47 RPKM
DRB1: 5.59 RPKM
DQA1: 0.44 RPKM
-----------4 digit typing results-------------
#Locus Allele 1 Confidence Allele 2 Confidence
!DQA DQA1*01:02' 0.1511134 DQA1*02:01 0.0
!DQB DQB1*02:01' 0.02321615 DQB1*05:01 0.42202
!DRB DRB1*15:02' 2.595144e-05 DRB1*07:01 0.321219