这是为了回答以前的一个疑问:任意给定基因组的 chr:pos, 判断它在哪个基因上面?这个程序难吗?
基因的chr,start,end都是已知的
学术一点讲述这个问题:已知CNV数据在染色体上的position如chr1:2075000-2930999,怎样批量获取其对应的Gene Symbol呢(批量)
数据如下:
head gene_position.hg19 //共21629行
1 chr19 58858171 58874214 A1BG ENSG00000121410
2 chr12 9220303 9268558 A2M ENSG00000175899
3 chr12 9381128 9386803 A2MP1 ENSG00000256069
9 chr8 18027970 18081198 NAT1 ENSG00000171428
10 chr8 18248754 18258723 NAT2 ENSG00000156006
12 chr14 95058394 95090390 ENSG00000273259
13 chr3 151531860 151546276 AADAC ENSG00000114771
14 chr2 219128851 219134893 AAMP ENSG00000127837
15 chr17 74449432 74466199 AANAT ENSG00000129673
16 chr16 70286296 70323412 AARS ENSG00000090861
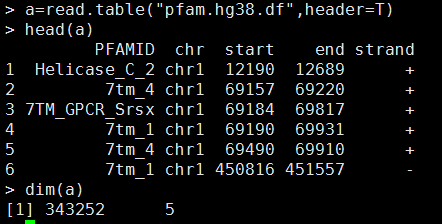
head pfam.df.hg19.bed //共340960行
chr1 12190 12689 Helicase_C_2 0 + 12190 12689 255,255,0
chr1 69157 69220 7tm_4 0 + 69157 69220 255,255,0
chr1 69184 69817 7TM_GPCR_Srsx 0 + 69184 69817 255,255,0
chr1 69190 69931 7tm_1 0 + 69190 69931 255,255,0
chr1 69490 69910 7tm_4 0 + 69490 69910 255,255,0
现在需要对我们的pfam数据进行注释,根据每一行的chr和pos来看看是属于哪一个基因
总共会有338879 条pfam记录可以注释上基因。
注释之后应该是 head pfam.gene.df.hg19 这个样子
CDK11B chr1 1571423 1573930 Pkinase 0 - 1571423 1573930 255,255,0
CDK11B chr1 1572048 1573921 Pkinase_Tyr 0 - 1572048 1573921 255,255,0
CDK11B chr1 1572120 1572823 Kinase-like 0 - 1572120 1572823 255,255,0
CDK11B chr1 1572120 1572820 Kinase-like 0 - 1572120 1572820 255,255,0
CDK11B chr1 1572120 1572817 Kinase-like 0 - 1572120 1572817 255,255,0
CDK11B chr1 1573173 1573918 Kinase-like 0 - 1573173 1573918 255,255,0
CDK11B chr1 1575747 1577317 Daxx 0 - 1575747 1577317 255,255,0
CDK11B chr1 1576417 1577347 Nop14 0 - 1576417 1577347 255,255,0
CDK11B chr1 1576423 1577332 Mitofilin 0 - 1576423 1577332 255,255,0
CDK11B chr1 1576432 1577317 SAPS 0 - 1576432 1577317 255,255,0
我的第一个程序用的是全基因位点扫描到hash的方法。这样需要扫描13,1390,4974个位点,多于三分之一的基因组,这样是非常浪费内存的,尤其是keys需要多个字节。
我用了256G的服务器都没有运行完。
后来我取巧了把我的gene_position.hg19文件用split命令分成了25个,然后循环25次对pfam.df.hg19.bed 文件进行注释。
这样的确可以解决问了,而且只需要32G的内存的服务器即可,时间也很快,就十多分钟吧。
但这只是取巧的方法,应该要从算法上面优化,首先我仅仅做一个改动,就是不再扫描全基因的位点,对每个基因,我以1K的窗口来取位点进行扫描。这样我判断pfam的坐标时候,也以1K为最小单位进行判断。
这样只需要不到30s就可以出结果,总共注释了303474条pfam记录,还不是最终的338879,因为我这次只注释了基因的1000整数倍基因区间,这样如果pfam记录落在一个基因的起始终止点不到1K位置时就不会被注释。这时候需要对代码进行继续优化。
脚步懒得上传了,在我的有道云笔记里面。
http://note.youdao.com/share/?id=58e66d138e9434284ffa61c53b65abdc&type=note