看到了一个2022的文章,标题是:《Bromodomain Inhibitors Modulate FcgR-Mediated Mononuclear Phagocyte Activation and Chemotaxis》。这个研究里面的转录组数据是通过RNA测序(RNA-seq)技术获得的,这些数据来自两个不同的细胞类型:树突细胞(Dendritic Cells, DCs)和巨噬细胞(Macrophages),以及它们在不同实验条件下的反应。 Continue reading
Monthly Archives: 9月 2024
让单细胞差异更加明显你需要调整配色方案
安排学徒复现一个新鲜出炉的阿兹海默症的单细胞文章:《Characterisation of premature cell senescence in Alzheimer’s disease using single nuclear transcriptomics》: Continue reading
欧美我来啦!
其实已经在朋友圈多次宣告过我要前往欧美,主要就是想拖家带口的换个环境生活,这个是最高目标。
这个也是我这么多年坚持把博士学位修完的唯一目的:拥有环游世界的旅居生活的主动权! Continue reading
你永远不知道公共数据集给你埋什么坑
不同格式的单细胞表达量矩阵文件读取的分门别类的代码我们都分享了好多次,最后都是要成为 Seurat对象。详见:读取不同格式的单细胞转录组数据及遇到问题的解决办法,简单的汇总一下就是: Continue reading
你的肿瘤单细胞肿瘤数据能区分这7种巨噬细胞吧
刷到了一个2022的巨噬细胞相关的单细胞综述文章:《Macrophage diversity in cancer revisited in the era of single-cell omics》,这篇综述文章深入探讨了肿瘤相关巨噬细胞(Tumor-Associated Macrophages, TAMs)的单细胞亚群,并根据其特征基因、富集通路及潜在功能,将TAMs分为7个不同的亚群。 Continue reading
可以每一条代谢通路都激活吗
最近刷到了2023发表在NC杂志的男性乳腺癌患者的单细胞转录组图谱文章,标题是:《Single-cell transcriptome analysis indicates fatty acid metabolism-mediated metastasis and immunosuppression in male breast cancer》
其中附件有一张图是男性和女性的乳腺癌患者肿瘤细胞表达量差异基因的代谢通路打分后的差异热图,如下所示: Continue reading
每个样品搞这么多细胞干什么啊(这样谁相信你的数据分析结论?)
交流群有小伙伴分享了一个新鲜出炉的2024的单细胞转录组配合空间单细胞的文献:《Single-cell and spatial transcriptomics analysis of non-small cell lung cancer》,可以看到这个研究的细胞数量还是很可观的,接近100万啦 : Continue reading
流式细胞都删不掉的亚群有什么特殊之处吗
前面我们分享了流式细胞这个技术在单细胞转录组课题的应用,详见:流式细胞筛选能保证多大程度的细胞亚群纯度呢,也就是说其实它并不能保证我们百分百获取的都是目标单细胞亚群从而对它进行细致的 探索。这一点也是大家总是在微信交流群提问,我才特意整理的: Continue reading
有点意思啊,两次都能定位到关键基因和通路!
我们这个月的马拉松生信入门课程进行到了表达量数据分析单元课,虽然说目前授课的案例主要还是基于20多年前的芯片技术的文章,但是数据分析的策略是一通百通的, 表达量矩阵可以来自于转录组测序技术,亦或是蛋白质组技术。 Continue reading
巨噬细胞极化的打分结果散点图
有小伙伴看了我们在单细胞天地公众号推荐的文献:多组学来量化原位和侵袭的肢端黑色素瘤差异后,发现了里面的免疫细胞的髓系免疫细胞里面的巨噬细胞的一个图没有意思,求组想知道如何分析拿到数据并且可视化。
如下所示的巨噬细胞极化的打分结果散点图: Continue reading
就离谱啊,是私藏一半的基因吗?
看到朋友圈好多人转发了一个最新的胃癌单细胞研究,数据集是GSE239676,在GEO界面可以看到如下所示的文件: Continue reading
间充质细胞的细分亚群情况
我在内皮细胞的细分亚群情况里面提到了,肿瘤微环境里面的stromal不仅仅是 fibro 和endo,还有周细胞和SMC,不过大多数情况下肿瘤样品里面的基质细胞并不多,所以不一定能细分清楚。其中内皮细胞主要是区分成为了淋巴内皮和血管内皮, 其中血管可以细分为动脉静脉和毛细血管: Continue reading
换一个技术工具结果就完全不同的RNA速率分析你还敢用吗
看到了一个预印本的文章:《Challenges and Progress in RNA Velocity: Comparative Analysis Across Multiple Biological Contexts,值得单细胞技术领域的小伙伴们深刻反思:因为测评后发现五大RNA速率计算方法结果迥异,这可能会对细胞状态动态的理解产生重要影响。文献详见 doi: https://doi.org/10.1101/2024.06.25.600667
单细胞转录组里面的RNA速率分析重要性 Continue reading
幻想破灭吧!这大环境还是得多做一手准备啊!
大清早刷朋友圈看到了有小伙伴截图分享了一个科研服务的“故事”or“事故”,如下所示:

这个问题其实聊到过很多次了,详见: Continue reading
花二十万做的大队列转录组告诉你不能这样设计课题啊!
学员在掌握了我们的授课的GEO数据挖掘之后通常是会试试看自己感兴趣的科研领域的表达量芯片或者转录组测序公开数据集,然后就各自碰壁,因为绝大部分文献在公开自己的数据的时候往往是会埋一些不大不小的坑。 Continue reading
关于r包安装到哪里了以及如何看自己的r包数量
好久以前(六年前吧),我录制了一个简单的r语言和rstudio软件安装视频,顺手让大家安装一些包试试看,代码如下所: Continue reading
发在cells的单细胞可视化网页工具你确定不了解一下吗
网页工具:《scViewer: An Interactive Single-Cell Gene Expression Visualization Tool》
详情见:Cells 2023, ; https://doi.org/10.3390/cells12111489 Continue reading
二十多万个细胞的单细胞数据集当然是继续顶啊
前面我在 十多万个细胞数量就顶不住了吗提到了一个关键的转换, 稀疏矩阵:
ct <- as.matrix(ct, type="dgCMatrix")
马上交流群就有小伙伴反馈了同款的bug,是二十多万个细胞的单细胞数据集,但是他自己就算是转换成为稀疏矩阵也没有解决问题。。。
# 参考:https://mp.weixin.qq.com/s/tw7lygmGDAbpzMTx57VvFw
ct=data.table::fread('GSE183852_Integrated_Counts.csv.gz',
data.table = F)
ct[1:4,1:4]
tail(ct[ ,1:4])
rownames(ct)=ct[,1]
ct=ct[,-1]
tmp <- as.matrix(ct, type="dgCMatrix")
sce.all=CreateSeuratObject(counts = tmp )
如下所示:
> ct[1:4,1:4]
gene H_ZC-11-292_TAAGTGCAGCAGGTCA H_ZC-11-292_ACAGCCGGTCATACTG
1 RP11-34P13.3 0 0
2 FAM138A 0 0
3 OR4F5 0 0
4 RP11-34P13.7 0 0
> tail(ct[ ,1:4])
gene H_ZC-11-292_TAAGTGCAGCAGGTCA
45063 AC136352.3 0
45064 AC136352.2 0
45065 AC171558.3 0
45066 BX004987.1 0
45067 AC145212.1 0
45068 MAFIP 0
Warning: Data is of class matrix. Coercing to dgCMatrix.
Error: vector memory exhausted (limit reached?)
我也确实是在我们的共享服务器(2024的共享服务器交个朋友福利价仍然是800)测试了,发现并没有问题。就继续沟通,发现交流群的小伙伴其实还没有使用我们的超大内存服务器,仅仅是在自己的电脑里面跑这个代码,所以失败完全是理所当然啊!!!
但是,问题还是得为他解决,上面的GSE183852数据集是有接近27万个细胞,所以绝大部分人普通电脑很难跑通这个环节,很正常。
我做了一个简单的磁盘中间环节避免这个内存bug,如下所示:
lapply(0:8, function(i){
# i=1
print(i)
kp= seq(1,ncol(ct)) %in% seq(i*30000 ,(i+1)*30000)
print(table(kp))
tmp= ct[,kp]
tmp=as.matrix( tmp ,
type="dgCMatrix")
save(tmp,file = paste(i,'tmp.Rdata'))
})
也就是说,GSE183852数据集是有接近27万个细胞,我把它拆分成为了9个独立的3万个细胞的表达量矩阵文件并且存储在硬盘里面。
然后就可以关闭R后重新打开,进行批量读取:
library(Seurat)
sceList = lapply(0:8, function(i){
# i=1
print(i)
load(file = paste(i,'tmp.Rdata'))
print(dim(tmp))
sce =CreateSeuratObject(counts = tmp,
#project = i ,
min.cells = 5,
min.features = 300 )
print(sce)
return(sce)
})
sce.all=merge(x=sceList[[1]],
y=sceList[ -1 ] )
names(sce.all@assays$RNA@layers)
sce.all[["RNA"]]$counts
# Alternate accessor function with the same result
LayerData(sce.all, assay = "RNA", layer = "counts")
sce.all <- JoinLayers(sce.all)
dim(sce.all[["RNA"]]$counts )
当然了,如果是你有服务器(2024的共享服务器交个朋友福利价仍然是800),就没必要上面的这样的如此曲折的分析了。
接下来就可以进行常规的降维聚类分群哈, 如下所示的初步结果:
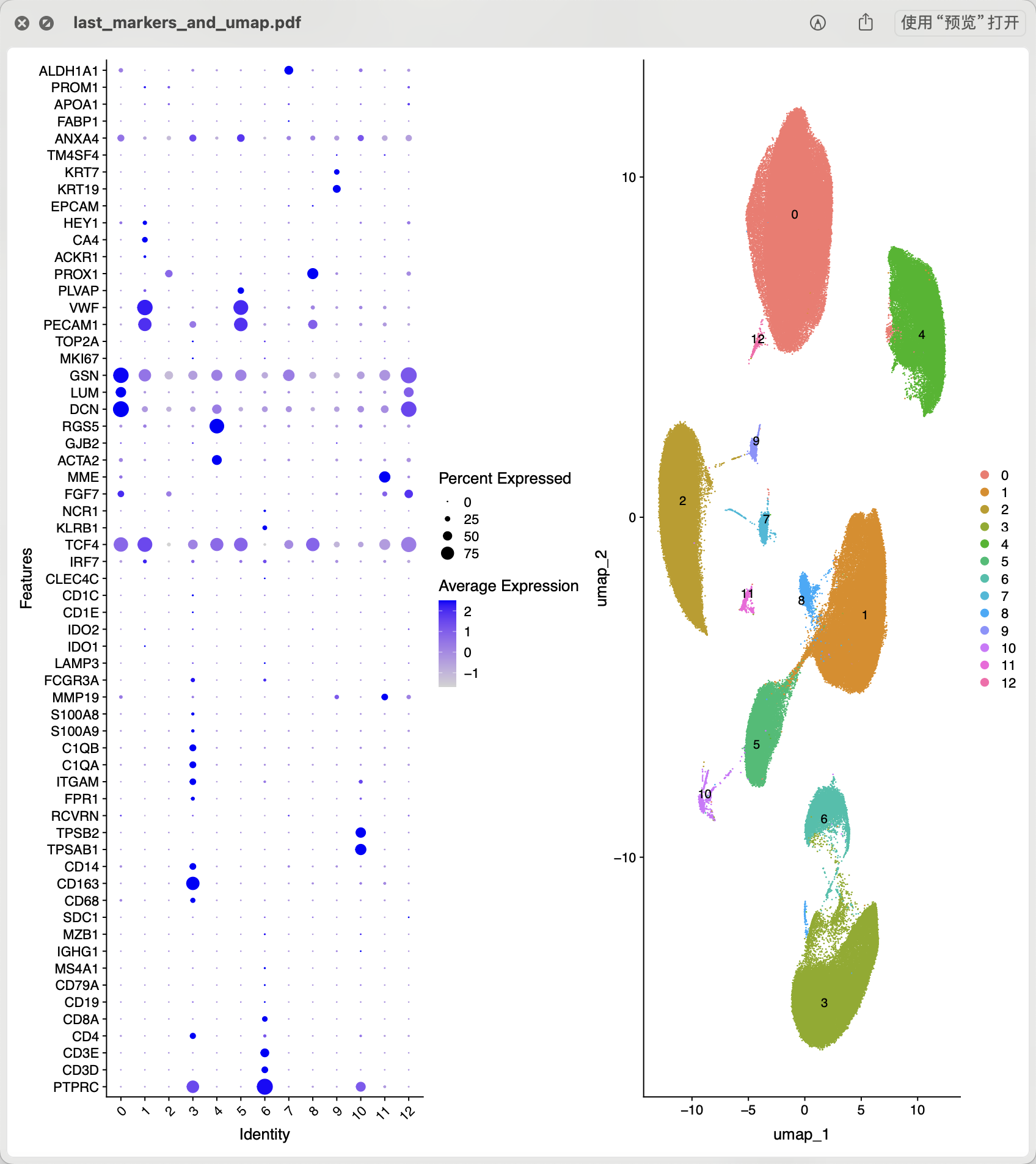
基本上可以看到下面的这些亚群是可以手动命名的:
celltype[celltype$ClusterID %in% c( 6 ),2]='lymphocytes'
celltype[celltype$ClusterID %in% c( 3 ),2]='myeloids'
celltype[celltype$ClusterID %in% c( 1,5 ),2]='endo'
celltype[celltype$ClusterID %in% c( 8 ),2]='L-endo'
celltype[celltype$ClusterID %in% c( 9 ),2]='epi'
celltype[celltype$ClusterID %in% c( 0 ),2]='fibro'
celltype[celltype$ClusterID %in% c( 4 ),2]='SMC'
celltype[celltype$ClusterID %in% c( 10 ),2]='Mast'
celltype[celltype$ClusterID %in% c( 12 ),2]='double'
然后需要看文章里面的基因列表:
补充进行可视化:
cg='RYR2 MYH11 DCN VWF CCL21 NRXN1 HAS1 KIT MZB1 PLIN1 C1QC KCNJ8 LEPR CD3E'
gene_list=trimws(strsplit(cg,' ')[[1]])
gene_list
p2 = DotPlot( sce.all.int, features = gene_list,
group.by = 'RNA_snn_res.0.1') +
theme(axis.text.x = element_text(angle = 45, vjust = 0.5, hjust=0.5))
p2
ggsave('heart-0.1.pdf',width=8)
就可以继续给名字
celltype[celltype$ClusterID %in% c(11 ),2]='Adipo'
celltype[celltype$ClusterID %in% c( 7 ),2]='Neuron'
celltype[celltype$ClusterID %in% c( 2 ),2]='CM'
最后的效果,我感觉我做的比文章要好一点,哈哈哈,如下所示:

学徒作业
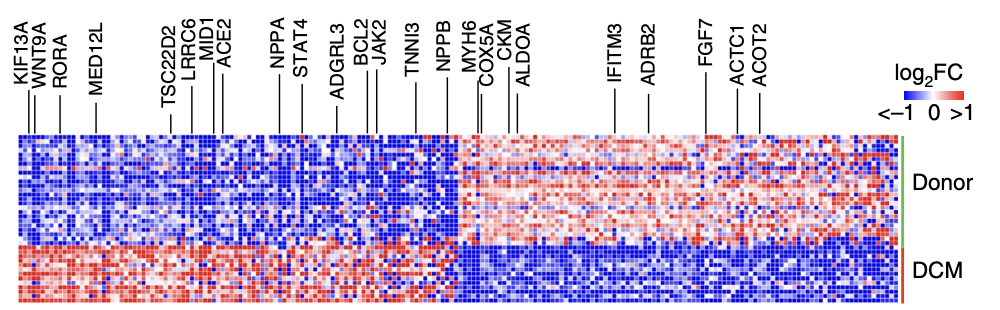
首先呢,完成上面的GSE183852数据集 接近27万个细胞的降维聚类分群和命名,然后读一下文章:《Single-cell transcriptomics reveals cell-type- specific diversification in human heart failure》,然后做一个简单的差异分析即可:
Differential expression analy- sis by pseudobulk and single-cell approaches across disease state revealed a large number of genes significantly upregulated (NPPA, NPPB, ACE2 and KIF13A) and downregulated (MYH6, ADRB2 and CKM) in DCM samples compared to non-diseased donors
并且绘制如下所示的差异基因的热图:
都2024了还有人在小红书找生信代做吗
天啊撸,朋友圈刷到了一个小伙伴分享了生物信息学项目委托事故,来源于小红书,聊天记录有12页,实在是太长了,所以就仅仅是截图一下苦主的单方面的事件汇总吧,如下所示: Continue reading
蛋白质组学公共数据库资源汇总
提到蛋白质数据库,大家都很熟悉了,基本上每个数据库都会配备一个网页工具让大家查询或者做一些简单的蛋白质数据分析,以下是一些知名的蛋白质组数据库: Continue reading