推荐5个生物信息学领域的教授
排名不分先后:
推荐宾夕法尼亚州立大学的一个教授Istvan Albert
关注一下华盛顿大学医学院的教授Obi L. Griffith
关注一下华盛顿大学医学院的教授Malachi Griffith
Linked In: Profile
Twitter: Feed
Google Scholar: Citations
Research Gate: Profile
Scopus: Profile
Open Research ID: Profile
Github: Profile
BioStar: Profile
SeqAnswers: Profile
Code Academy: Profile
Iterative Genomics Consulting: Company website
Flickr: Photostream
www.dgidb.org
www.alexaplatform.org
关注一下麦吉尔大学的Pablo Cingolani教授
推荐弗吉尼亚大学的stephen教授
关于qsub和condor两种在集群上面提交任务的方式比对
毕竟不是计算机科班出身,很多计算机概念我其实并不清楚,很多时候对一个任务的解决只是凭着自己的悟性来模仿接触到的做法,大多数情况下并无可厚非,能完成任务即可,但是碰到多人协作的环境,往往就出了问题。
而condor其实很少见的。
condor_q 可以用来查看任务提交情况
任务提交模式还是挺有用的
一个表达芯片数据处理实例
RNA-seq流程需要进化啦!
Tophat 首次被发表已经是6年前
Cufflinks也是五年前的事情了
Star的比对速度是tophat的50倍,hisat更是star的1.2倍。
stringTie的组装速度是cufflinks的25倍,但是内存消耗却不到其一半。
Ballgown在差异分析方面比cuffdiff更高的特异性及准确性,且时间消耗不到cuffdiff的千分之一
Bowtie2+eXpress做质量控制优于tophat2+cufflinks和bowtie2+RSEM
Sailfish更是跳过了比对的步骤,直接进行kmer计数来做QC,特异性及准确性都还行,但是速度提高了25倍
kallisto同样不需要比对,速度比sailfish还要提高5倍!!!
参考:https://speakerdeck.com/stephenturner/rna-seq-qc-and-data-analysis-using-the-tuxedo-suite
Shell里面的各种括号的区别
[],[[]],(),(()),{},{{}},以及在前面加上$的区别,以及它们互相杂交组合的区别!!!
[[ ]] double brackets
(())Double parentheses
{{}}double curly brackets
我们必须要记住的是下面
[] 相当于test,作逻辑判断
$( ) 与` ` (反引号) 都是用来做命令替换用
${ } 吧... 它其实就是用来作变量替换用的啦
(())就是用来计算的,相当于expr函数。
http://tldp.org/LDP/abs/html/index.html
我们首先看看一对的括号
首先[]是用来逻辑判断的,必须有空格
if [ -f binom.py ]
then
echo 'binom.py exists'
fi
或者
nub=$((i%4))
#echo $nub
if [ $nub == 0 ];then
echo "we need to sleep 4 hours"
sleep 14000
fi
这个[]操作符等价于test函数
if test $1 -gt 0
then
echo "$1 number is positive"
fi
但是都必须有空格!!!
参考:http://www.freeos.com/guides/lsst/ch03sec02.html
关于shell的test操作符还有很多http://tldp.org/LDP/abs/html/fto.html
( ) 将command group 置于 sub-shell 去执行,也称 nested sub-shell。
{ } 则是在同一个 shell 内完成,也称为non-named command group。
补充一个: {} 还可以做变量扩展 {5..9} 或者 {abcd}e, 自己运行一下就知道效果啦
这两个差异很小,而且一般用不着,就不讲了。
那么这一对的括号加上了$符号后又变成了上面鬼东西呢?
当然,只有:$( ) 与${ }才是合法的。
在 bash shell 中,$( ) 与` ` (反引号) 都是用来做命令替换用(command substitution)的。
在操作上,用$( ) 或` ` 都无所谓,用$( )的优点是:
1, ` ` 很容易与' ' ( 单引号)搞混乱,尤其对初学者来说
2, 在多层次的复合替换中,` ` 须要额外的跳脱( \` )处理,而$( ) 则比较直观
再让我们看${ } 吧... 它其实就是用来作变量替换用的啦。
一般情况下,$var 与${var} 并没有啥不一样。
但是用${ } 会比较精确的界定变量名称的范围,比方说:
[code][/code]
$ A=B
$ echo $AB
还可以用来截取变量,这个就很多花样啦
# 是去掉左边(在鉴盘上# 在$ 之左边)
% 是去掉右边(在鉴盘上% 在$ 之右边)
单一符号是最小匹配﹔两个符号是最大匹配
然后我们看看两对的括号:
nub=$((i%4)) 等价于$nub=`expr $i % 1` ;
((i++)) 等价于$i=`expr $i + 1` ;
所以(())就是用来计算的,而且里面的变量不需要$来标记啦
(在 $(( )) 中的变量名称,可于其前面加$ 符号来替换,也可以不用)
在(())前面加上$只是为了把计算结果给保存而已。
而两个中括号和两个大括号都是不合法的!
用 GMAP/GSNAP软件进行RNA-seq的alignment
软件的解说ppt :http://www.mi.fu-berlin.de/wiki/pub/ABI/CompMethodsWS11/MHuska_GSNAP.pdf
用freebayes来call snps
wget http://clavius.bc.edu/~erik/freebayes/freebayes-5d5b8ac0.tar.gz
tar xzvf freebayes-5d5b8ac0.tar.gz
cd freebayes
make
一个小插曲,安装的过程报错:/bin/sh: 1: cmake: not found
所以我需要自己下载安装cmake,然后把cmake添加到环境变量
首先下载源码包http://www.cmake.org/cmake/resources/software.html
wget http://cmake.org/files/v3.3/cmake-3.3.2.tar.gz
解压进去,然后源码安装三部曲,首先 ./configu 然后make 最后make install
cmake 会默认安装在 /usr/local/bin 下面,这里需要修改,因为你可能没有 /usr/local/bin 权限,安装到自己的目录,然后把它添加到环境变量!
step2:准备数据
wget http://bioinformatics.bc.edu/marthlab/download/gkno-cshl-2013/chr20.fawget ftp://ftp.ncbi.nlm.nih.gov/1000genomes/ftp/data/NA12878/alignment/NA12878.chrom20.ILLUMINA.bwa.CEU.low_coverage.20121211.bam
wget ftp://ftp.ncbi.nlm.nih.gov/1000genomes/ftp/data/NA12878/alignment/NA12878.chrom20.ILLUMINA.bwa.CEU.low_coverage.20121211.bam.bai用这个代码就可以下载千人基因组计划的NA12878样本的第20号染色体相关数据啦
freebayes -f chr20.fa \
NA12878.chrom20.ILLUMINA.bwa.CEU.low_coverage.20121211.bam >NA12878.chr20.freebayes.vcf一般就用默认参数即可
broad_institute收集的癌症数据
| 肾上腺皮质 | Adrenocortical carcinoma | ACC | 92 | Browse | Browse |
| 膀胱,尿路上皮 | Bladder urothelial carcinoma | BLCA | 412 | Browse | Browse |
| 乳腺癌 | Breast invasive carcinoma | BRCA | 1098 | Browse | Browse |
| 子宫颈 | Cervical and endocervical cancers | CESC | 307 | Browse | Browse |
| 胆管癌 | Cholangiocarcinoma | CHOL | 36 | Browse | Browse |
| 结肠腺癌 | Colon adenocarcinoma | COAD | 460 | Browse | Browse |
| 大肠腺癌 | Colorectal adenocarcinoma | COADREAD | 631 | Browse | Browse |
| 淋巴肿瘤弥漫性大B细胞淋巴瘤 | Lymphoid Neoplasm Diffuse Large B-cell Lymphoma | DLBC | 58 | Browse | Browse |
| 食管 | Esophageal carcinoma | ESCA | 185 | Browse | Browse |
| FFPE试点二期 | FFPE Pilot Phase II | FPPP | 38 | None | Browse |
| 胶质母细胞瘤 | Glioblastoma multiforme | GBM | 613 | Browse | Browse |
| 脑胶质瘤 | Glioma | GBMLGG | 1129 | Browse | Browse |
| 头颈部鳞状细胞癌 | Head and Neck squamous cell carcinoma | HNSC | 528 | Browse | Browse |
| 肾嫌色 | Kidney Chromophobe | KICH | 113 | Browse | Browse |
| 泛肾 | Pan-kidney cohort (KICH+KIRC+KIRP) | KIPAN | 973 | Browse | Browse |
| 肾透明细胞癌 | Kidney renal clear cell carcinoma | KIRC | 537 | Browse | Browse |
| 肾乳头细胞癌 | Kidney renal papillary cell carcinoma | KIRP | 323 | Browse | Browse |
| 急性髓系白血病 | Acute Myeloid Leukemia | LAML | 200 | Browse | Browse |
| 脑低级神经胶质瘤 | Brain Lower Grade Glioma | LGG | 516 | Browse | Browse |
| 肝癌 | Liver hepatocellular carcinoma | LIHC | 377 | Browse | Browse |
| 肺腺癌 | Lung adenocarcinoma | LUAD | 585 | Browse | Browse |
| 肺鳞状细胞癌 | Lung squamous cell carcinoma | LUSC | 504 | Browse | Browse |
| 间皮瘤 | Mesothelioma | MESO | 87 | Browse | Browse |
| 卵巢浆液性囊腺癌 | Ovarian serous cystadenocarcinoma | OV | 602 | Browse | Browse |
| 胰腺癌 | Pancreatic adenocarcinoma | PAAD | 185 | Browse | Browse |
| 嗜铬细胞瘤和副神经节瘤 | Pheochromocytoma and Paraganglioma | PCPG | 179 | Browse | Browse |
| 前列腺癌 | Prostate adenocarcinoma | PRAD | 499 | Browse | Browse |
| 直肠腺癌 | Rectum adenocarcinoma | READ | 171 | Browse | Browse |
| 肉瘤 | Sarcoma | SARC | 260 | Browse | Browse |
| 皮肤皮肤黑色素瘤 | Skin Cutaneous Melanoma | SKCM | 470 | Browse | Browse |
| 胃腺癌 | Stomach adenocarcinoma | STAD | 443 | Browse | Browse |
| 胃和食管癌 | Stomach and Esophageal carcinoma | STES | 628 | Browse | Browse |
| 睾丸生殖细胞肿瘤 | Testicular Germ Cell Tumors | TGCT | 150 | Browse | Browse |
| 甲状腺癌 | Thyroid carcinoma | THCA | 503 | Browse | Browse |
| 胸腺瘤 | Thymoma | THYM | 124 | Browse | Browse |
| 子宫内膜癌 | Uterine Corpus Endometrial Carcinoma | UCEC | 560 | Browse | Browse |
| 子宫癌肉瘤 | Uterine Carcinosarcoma | UCS | 57 | Browse | Browse |
| 葡萄膜黑色素瘤 | Uveal Melanoma | UVM | 80 | Browse | Browse |
看起来癌症很多呀,任重道远
perl的模块组织方式
如何使用自己写的私人模块
/www/modules/MyMods/Foo.pm
/www/modules/MyMods/Bar.pmAnd then where I use them:
use lib qw(/www/modules);useMyMods::Foo; useMyMods::Bar;
As reported by "perldoc -f use":
It is exactly equivalent to
BEGIN { require Module; import Module LIST; }
except that Module must be a bareword.
Putting that another way, "use" is equivalent to:
- running at compile time,
- converting the package name to a file name,
require-ing that file name, andimport-ing that package.
So, instead of calling use, you can call require and import inside a BEGIN block:
BEGIN{require'../EPMS.pm';
EPMS->import();}And of course, if your module don't actually do any symbol exporting or other initialization when you call import, you can leave that line out:
BEGIN{require'../EPMS.pm';}一个基因坐标定位到具体基因的程序的改进
这是为了回答以前的一个疑问:任意给定基因组的 chr:pos, 判断它在哪个基因上面?这个程序难吗?
基因的chr,start,end都是已知的
学术一点讲述这个问题:已知CNV数据在染色体上的position如chr1:2075000-2930999,怎样批量获取其对应的Gene Symbol呢(批量)
数据如下:
head gene_position.hg19 //共21629行
1 chr19 58858171 58874214 A1BG ENSG00000121410
2 chr12 9220303 9268558 A2M ENSG00000175899
3 chr12 9381128 9386803 A2MP1 ENSG00000256069
9 chr8 18027970 18081198 NAT1 ENSG00000171428
10 chr8 18248754 18258723 NAT2 ENSG00000156006
12 chr14 95058394 95090390 ENSG00000273259
13 chr3 151531860 151546276 AADAC ENSG00000114771
14 chr2 219128851 219134893 AAMP ENSG00000127837
15 chr17 74449432 74466199 AANAT ENSG00000129673
16 chr16 70286296 70323412 AARS ENSG00000090861
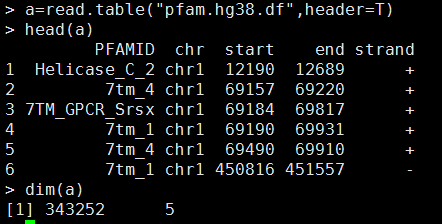
head pfam.df.hg19.bed //共340960行
chr1 12190 12689 Helicase_C_2 0 + 12190 12689 255,255,0
chr1 69157 69220 7tm_4 0 + 69157 69220 255,255,0
chr1 69184 69817 7TM_GPCR_Srsx 0 + 69184 69817 255,255,0
chr1 69190 69931 7tm_1 0 + 69190 69931 255,255,0
chr1 69490 69910 7tm_4 0 + 69490 69910 255,255,0
现在需要对我们的pfam数据进行注释,根据每一行的chr和pos来看看是属于哪一个基因
总共会有338879 条pfam记录可以注释上基因。
注释之后应该是 head pfam.gene.df.hg19 这个样子
CDK11B chr1 1571423 1573930 Pkinase 0 - 1571423 1573930 255,255,0
CDK11B chr1 1572048 1573921 Pkinase_Tyr 0 - 1572048 1573921 255,255,0
CDK11B chr1 1572120 1572823 Kinase-like 0 - 1572120 1572823 255,255,0
CDK11B chr1 1572120 1572820 Kinase-like 0 - 1572120 1572820 255,255,0
CDK11B chr1 1572120 1572817 Kinase-like 0 - 1572120 1572817 255,255,0
CDK11B chr1 1573173 1573918 Kinase-like 0 - 1573173 1573918 255,255,0
CDK11B chr1 1575747 1577317 Daxx 0 - 1575747 1577317 255,255,0
CDK11B chr1 1576417 1577347 Nop14 0 - 1576417 1577347 255,255,0
CDK11B chr1 1576423 1577332 Mitofilin 0 - 1576423 1577332 255,255,0
CDK11B chr1 1576432 1577317 SAPS 0 - 1576432 1577317 255,255,0
我的第一个程序用的是全基因位点扫描到hash的方法。这样需要扫描13,1390,4974个位点,多于三分之一的基因组,这样是非常浪费内存的,尤其是keys需要多个字节。
我用了256G的服务器都没有运行完。
后来我取巧了把我的gene_position.hg19文件用split命令分成了25个,然后循环25次对pfam.df.hg19.bed 文件进行注释。
这样的确可以解决问了,而且只需要32G的内存的服务器即可,时间也很快,就十多分钟吧。
但这只是取巧的方法,应该要从算法上面优化,首先我仅仅做一个改动,就是不再扫描全基因的位点,对每个基因,我以1K的窗口来取位点进行扫描。这样我判断pfam的坐标时候,也以1K为最小单位进行判断。
这样只需要不到30s就可以出结果,总共注释了303474条pfam记录,还不是最终的338879,因为我这次只注释了基因的1000整数倍基因区间,这样如果pfam记录落在一个基因的起始终止点不到1K位置时就不会被注释。这时候需要对代码进行继续优化。
脚步懒得上传了,在我的有道云笔记里面。
http://note.youdao.com/share/?id=58e66d138e9434284ffa61c53b65abdc&type=note
affymetix的基因表达芯片数据差异基因分析
我主要是看了一个差异分析的教程,讲的非常详细,全面,我先简单列出这个教程,然后再贴出我的代码
基因芯片(Affymetrix)分析2:芯片数据预处理
基因芯片(Affymetrix)分析3:获取差异表达基因
基因芯片(Affymetrix)分析4:GO和KEGG分析
基因芯片(Affymetrix)分析5:聚类分析
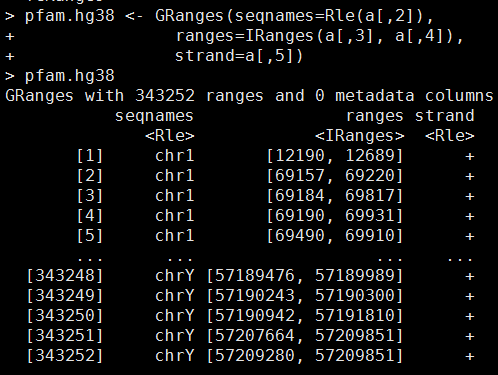
liftover基因组版本直接的coordinate转换
chr7 127471196 127472363 Pos1 0 + 127471196 127472363 255,0,0 chr7 127472363 127473530 Pos2 0 + 127472363 127473530 255,0,0
library(GenomicRanges)
pfam.hg38 <- GRanges(seqnames=Rle(a[,2]),
ranges=IRanges(a[,3], a[,4]),
strand=a[,5])
library(rtracklayer)
ch = import.chain("hg38ToHg19.over.chain")
pfam.hg19 = liftOver(pfam.hg38, ch)
JQuery学习笔记
以后写这样的文章就直接用有道云笔记分享啦,这样可以节约这个免费的云服务器的空间。
jquery学习笔记第一弹:基础语法
http://note.youdao.com/share/?id=82021515144eb4820762e9fdbc686340&type=note
JQuery笔记第二弹:ppt效果操作
http://note.youdao.com/share/?id=08eb606b2084b9b0d8c9eb5ef72e3433&type=note
JQuery笔记第三弹:操作html元素
http://note.youdao.com/share/?id=fb8ff7deeb186adb82751838bf82cfbe&type=note
JQuery笔记第四弹:循环,遍历,判断等语句实现
http://note.youdao.com/share/?id=746ac6f1a801351f49d13cb3d7a335bf&type=note
JQuery笔记第五弹:Ajax实现
http://note.youdao.com/share/?id=0b2c6fb8c89e307ec79602e6d67e7c66&type=note
JQuery参考手册-函数大全
http://note.youdao.com/share/?id=2e926f98c9bd51b1192d309706f8c1ca&type=note
生物统计学习资料大全
http://stat.ethz.ch/education/semesters/ss2015
2012-LAD的三个亚型的不同生物学意义
文献名:Differential Pathogenesis of Lung Adenocarcinoma Subtypes Involving Sequence Mutations, Copy Number, Chromosomal Instability, and Methylation
Lung adenocarcinoma (LAD)的遗传变异度很大。
这个癌症可以分成三类:The LAD molecular subtypes (Bronchioid, Magnoid, and Squamoid)
然后我们在三个subtypes里面分析了以下四个特征,发现不同subtypes差异非常显著。
1、Gene mutation rates (EGFR, KRAS, STK11, TP53),
2、chromosomal instability,
3、regional copy number
4、genomewide DNA methylation
另外三个临床特征也是很显著。
1、Patient overall survival,
2、cisplatin plus vinorelbine therapy response
3、predicted gefitinib sensitivity
所以,我们的分类非常好,而且对临床非常有帮助。
对LAD的研究数据包括
1,DNA copy number
2,gene sequence mutation
3,DNA methylation
4,gene expression
即使是TP53这样的基因在LAD的突变率也才35%,所以我们的LAD应该更加细分,因为EGFR mutation and KRAS mutation这样的突变对治疗很有指导意义,细分更加有助于临床针对性治疗方案的选择。
我们选取了116个LAD样本的数据,分析了1,genome-wide gene expression,,2,genomewide DNA copy number, 3,genome-wide DNA methylation, 4,selected gene sequence mutations
得到的结论是:LAD molecular subtypes correlate with grossly distinct genomic alterations and patient therapy response
数据来源如下:
Gene expression --> Agilent 44 K microarrays.
DNA copy number --> Affymetrix 250 K Sty and SNP6 microarrays.
DNA methylation --> MSNP microarray assay.
DNA from EGFR, KRAS, STK11 and TP53 exons --> ABI sequencers
我们用的是R语言包 ConsensusClusterPlus根据gene expression 来对我们的LAD进行分类molecular subtypes
分类的基因有506个(the top 25% most variable genes, 3,045, using ConsensusClusterPlus),A nearest centroid subtype predictor utilizing 506 genes
这三类LAD的过表达基因参与不同的生物功能,
Bronchioid – excretion genes, asthma genes, and surfactants (SFTPB, SFTPC, SFTPD);
Magnoid – DNA repair genes, such as thymine-DNA glycosylase (TDG);
Squamoid – defense response genes, such as chemokine ligand 10 (CXCL10)
而且也对应不同的临床数据
Bronchioid had the most females, nonsmokers, early stage tumors, and low grade tumors, the greatest acinar content, the least necrosis, and the least invasion.
Squamoid had the most high grade tumors, the greatest solid content, and the lowest papillary content.
Magnoid had themost smokers and the heaviest smokers by pack years.
它们的基因突变pattern也有很大区别。
Bronchioid had the greatest EGFR mutation frequency
Magnoid had the greatest mutation frequencies in TP53, KRAS and STK11.
为了研究不同亚型癌症的突变模式的不同(genomewide mutation rates),我们同时又研究了a large set of rarely mutated genes (n = 623) from the Ding et al. cohort
结论:
Bronchioid subtype 更有可能受益于EGFR inhibitory therapy
Magnoid tumors also have severe genomic alterations including the greatest CIN, the most regional CN alterations, DNA hypermethylation, and the greatest genomewide mutation rate.
the Squamoid subtype displayed the fewest distinctive alterations that included only regional CN alterations