厦门大学不再提供R语言镜像
)
)
ubuntu对生信菜鸟来说是最好用的linux服务器,没有之一,因为它有apt-get。
1、JDK官网上http://www.oracle.com/technetwork/java/javase/downloads/index.html选择:
但是,如果你的服务器是64位的,请不要选择i586,选择你自己的机器对应的!
2、将打开终端,建立目录:
Sudo mkdir /usr/lib/java
3、将下载的 jdk-7u3-linux-i586.tar.gz移到这个文件夹下面并进行解压,改名字:
sudo mv jdk-7u3-linux-i586.tar.gz /usr/lib/java
sudo tar –xvf jdk-7u3-linux-i586.tar.gz
mv jdk1.7.0_03java-7-sun
4、修改环境变量:
在终端输入:vim /etc/profile
然后添加以下代码:
export JAVA_HOME=/usr/lib/java/jdk1.8.0_25
export JRE_HOME=${JAVA_HOME}/jre
export CLASSPATH=.:${JAVA_HOME}/lib:${JRE_HOME}/lib
export PATH=${JAVA_HOME}/bin:$PATH
保存之后,再运行下面命令更新电脑的配置文件
source /etc/profile 这个千万要记得!!!!
5、在终端中输入 java –version,显示:
jeydragon@jeydragon-VirtualBox:~$ java -version
java version "1.7.0_03"
Java(TM) SE Runtime Environment (build 1.7.0_03-b04)
Java HotSpot(TM) Client VM (build 22.1-b02, mixed mode)
表示安装成功
这个工具主要是针对不会bioperl不会API调取数据的生信纯菜鸟准备的,主要是方便大家批量研究某些感兴趣的基因,需要准备的数据就是基因名或者基因的ID号,能从该网站获取的资料非常多,可以是关于你的输入的基因名的各种数据库有的信息。
http://www.ensembl.org/biomart/
第一步:选取数据库,我一般选取人的
第二步,选择上传数据的格式
这个下拉框里面可以选取很多种格式,你随便张贴进去哪一种格式的基因ID都可以,也可以把做好的ID文件上传进去,批量获取基因信息。
我这里输入的是几个免疫基因。
第三步,选择下载数据的格式
首先可以选择你上传的gene的可以转换的各种ID
然后可以选择你上传的gene的各种序列
可以选择的信息非常多,基本上可以想到的转换在这里都能做!!!
但是,始终没有脚本方便,只适合不太懂编程的菜鸟使用!
然后点击result即可,看到结果还可以导出成txt文档,点击右上角的GO即可
之所以要讲一下这个,是因为trinity这个软件居然需要perl的模块才能使用,所以我必须自己安装几个模块。此教程可能过期了,请直接看最新版(perl模块安装大全)
前提是要有root权限,否则只能自己下载perl模块自己解压安装了。 Continue reading
刚买了一个空的云服务器,所以就试用体验了一下!
一.硬盘容量,系统盘很小,就30个G,但是有一个1T的空盘,自己格式化安装并挂载即可。
二.内存状况,11G
三。Cup数量,12核
四.磁盘文件状况
五.开通其它账号
adduser
六.服务器其它信息
七.测试下载速度
八.磁盘分区管理
首先看到的是32.2GB的系统盘,是标示符是xvda盘,被分成了三个区
然后是一个1T的硬盘,需要分区然后挂载
我分区后,分割成两个盘,一个给各个用户,还有一个放公共数据
apt-get install nfs-common
mkfs -t ext4 /dev/xvdd
mkfs.ext4 /dev/xvdd1
mount -t ext4 /dev/xvdd1 /home/
mount -t ext4 /dev/xvdd2 /data
九.脚本环境
This is perl 5, version 18, subversion 2 (v5.18.2)
/usr/lib/python2.7/site.pyc matches /usr/lib/python2.7/site.py
十.库文件状况
apt-get install unzip
apt-get install make
apt-get install gcc
十一.软件安装状况
常用生物信息学软件都可以自己安装,并且可以使用。
这个软件其实我真心不需要讲些什么了,它的官网写的太好了,简直就是软件说明书的典范
http://www.nipgr.res.in/ngsqctoolkit.html
它列出了它的几个功能模块,还给出了下载地址,还给出了说明文档,下载压缩包,解压即可使用啦
更重要的是给出了测试数据和测试的结果,而且还专门测试了不同测序平台及不同的测序策略的使用说明
里面就是一些perl测序,其实自己都可以写的,分成了四大类。
其中统计的那个平均测序质量,我在前面仿写fastqc就写过,至于那个统计N50,更是生信常用的脚本。
但是大家可以看看这个perl程序来学perl语言,蛮不错的这些程序,都写的很标准。
比如那个TrimmingReads.pl
可以根据四个参数来选择性的对我们的原始reads进行过滤,当然很多其它的程序也有类似的功能,它的参数分别是铲掉5端的几个碱基或者3端的,或者根据测序质量来切除碱基,或者根据reads长度来取舍,都是挺实用的功能。但是我一般用LengthSort和DynamicTrim那两个程序,原因很简单,我老师是这样用的,所以我习惯了,哈哈
一、下载安装该软件。
网上可以搜索到下载地址,解压之后make即可
一般都会报错
In file included from bam_cat.c:41:0:
htslib-1.1/htslib/bgzf.h:34:18: fatal error: zlib.h: No such file or directory
#include <zlib.h>
^
compilation terminated.
make: *** [bam_cat.o] Error 1
然后,居然就通过了,晕。有时候我实在是搞不定linux系统一些具体的原理,但是反正就是能用!学会搜索,学会试错即可。
直到两年后我才理解(linux下 的软件安装需要指定路径,而且是自己有权限的路径,2016年11月23日10:12:11),比如安装下面的方式来安装软件:
mkdir -p ~/biosoft/myBin
echo 'export PATH=/home/jianmingzeng/biosoft/myBin/bin:$PATH' >>~/.bashrc
source ~/.bashrc
cd ~/biosoft
mkdir cmake && cd cmake
wget http://cmake.org/files/v3.3/cmake-3.3.2.tar.gz
tar xvfz cmake-3.3.2.tar.gz
cd cmake-3.3.2
./configure --prefix=/home/jianmingzeng/biosoft/myBin ## 这里非常重要
make
make install
但是有些电脑会报另外一个错
#include <curses.h>
^
compilation terminated.
make: *** [bam_tview_curses.o] Error 1
我也顺便解决一下,因为以前我的服务器遇到过,也是很纠结的。
sudo apt-get install libncurses5-dev
二.准备数据及使用,见我的snp-caling流程
http://www.bio-info-trainee.com/?p=439
samtools view -bS tmp1.sam > tmp1.bam
samtools sort tmp1.bam tmp1.sorted
samtools index tmp1.sorted.bam
samtools mpileup -d 1000 -gSDf ../../../ref-database/hg19.fa tmp1.sorted.bam |bcftools view -cvNg – >tmp1.vcf
因为这个软件都是与bwa和bowtie等能产生sam文件的软件合作才能使用。
其中这个软件参数还是蛮多的,但是常用的就那么几个,网上也很容易找到教程
简单附上一点资料
samtools是一个用于操作sam和bam文件的工具合集。包含有许多命令。以下是常用命令的介绍
view命令的主要功能是:将sam文件转换成bam文件;然后对bam文件进行各种操作,比如数据的排序(不属于本命令的功能)和提取(这些操作是对bam文件进行的,因而当输入为sam文件的时候,不能进行该操作);最后将排序或提取得到的数据输出为bam或sam(默认的)格式。
bam文件优点:bam文件为二进制文件,占用的磁盘空间比sam文本文件小;利用bam二进制文件的运算速度快。
view命令中,对sam文件头部的输入(-t或-T)和输出(-h)是单独的一些参数来控制的。
Usage: samtools view [options] <in.bam>|<in.sam> [region1 [...]]默认情况下不加 region,则是输出所有的 region. Options:
-b output BAM 默认下输出是 SAM 格式文件,该参数设置输出 BAM 格式 -h print header for the SAM output 默认下输出的 sam 格式文件不带 header,该参数设定输出sam文件时带 header 信息 -H print header only (no alignments) -S input is SAM 默认下输入是 BAM 文件,若是输入是 SAM 文件,则最好加该参数,否则有时候会报错。
例子:
#将sam文件转换成bam文件$ samtools view -bS abc.sam > abc.bam$ samtools view -b -S abc.sam -o abc.bam
#提取比对到参考序列上的比对结果$ samtools view -bF 4 abc.bam > abc.F.bam #提取paired reads中两条reads都比对到参考序列上的比对结果,只需要把两个4+8的值12作为过滤参数即可$ samtools view -bF 12 abc.bam > abc.F12.bam #提取没有比对到参考序列上的比对结果$ samtools view -bf 4 abc.bam > abc.f.bam #提取bam文件中比对到caffold1上的比对结果,并保存到sam文件格式$ samtools view abc.bam scaffold1 > scaffold1.sam #提取scaffold1上能比对到30k到100k区域的比对结果$ samtools view abc.bam scaffold1:30000-100000 > scaffold1_30k-100k.sam #根据fasta文件,将 header 加入到 sam 或 bam 文件中$ samtools view -T genome.fasta -h scaffold1.sam > scaffold1.h.sam
sort对bam文件进行排序。
Usage: samtools sort [-n] [-m <maxMem>] <in.bam> <out.prefix> -m 参数默认下是 500,000,000 即500M(不支持K,M,G等缩写)。对于处理大数据时,如果内存够用,则设置大点的值,以节约时间。-n 设定排序方式按short reads的ID排序。默认下是按序列在fasta文件中的顺序(即header)和序列从左往右的位点排序。
例子:
$ samtools sort abc.bam abc.sort$ samtools view abc.sort.bam | less -S
将2个或2个以上的已经sort了的bam文件融合成一个bam文件。融合后的文件不需要则是已经sort过了的。
Usage: samtools merge [-nr] [-h inh.sam] <out.bam> <in1.bam> <in2.bam>[...] Options: -n sort by read names -r attach RG tag (inferred from file names) -u uncompressed BAM output -f overwrite the output BAM if exist -1 compress level 1 -R STR merge file in the specified region STR [all] -h FILE copy the header in FILE to <out.bam> [in1.bam] Note: Samtools' merge does not reconstruct the @RG dictionary in the header. Users must provide the correct header with -h, or uses Picard which properly maintains the header dictionary in merging.
必须对bam文件进行默认情况下的排序后,才能进行index。否则会报错。
建立索引后将产生后缀为.bai的文件,用于快速的随机处理。很多情况下需要有bai文件的存在,特别是显示序列比对情况下。比如samtool的tview命令就需要;gbrowse2显示reads的比对图形的时候也需要。
Usage: samtools index <in.bam> [out.index]
例子:
#以下两种命令结果一样$ samtools index abc.sort.bam$ samtools index abc.sort.bam abc.sort.bam.bai
对fasta文件建立索引,生成的索引文件以.fai后缀结尾。该命令也能依据索引文件快速提取fasta文件中的某一条(子)序列
Usage: samtools faidx <in.bam> [ [...]] 对基因组文件建立索引$ samtools faidx genome.fasta#生成了索引文件genome.fasta.fai,是一个文本文件,分成了5列。第一列是子序列的名称;第二列是子序列的长度;个人认为“第三列是序列所在的位置”,因为该数字从上往下逐渐变大,最后的数字是genome.fasta文件的大小;第4和5列不知是啥意思。于是通过此文件,可以定位子序列在fasta文件在磁盘上的存放位置,直接快速调出子序列。 #由于有索引文件,可以使用以下命令很快从基因组中提取到fasta格式的子序列$ samtools faidx genome.fasta scffold_10 > scaffold_10.fasta
tview能直观的显示出reads比对基因组的情况,和基因组浏览器有点类似。
Usage: samtools tview <aln.bam> [ref.fasta] 当给出参考基因组的时候,会在第一排显示参考基因组的序列,否则,第一排全用N表示。按下 g ,则提示输入要到达基因组的某一个位点。例子“scaffold_10:1000"表示到达第10号scaffold的第1000个碱基位点处。使用H(左)J(上)K(下)L(右)移动显示界面。大写字母移动快,小写字母移动慢。使用空格建向左快速移动(和 L 类似),使用Backspace键向左快速移动(和 H 类似)。Ctrl+H 向左移动1kb碱基距离; Ctrl+L 向右移动1kb碱基距离可以用颜色标注比对质量,碱基质量,核苷酸等。30~40的碱基质量或比对质量使用白色表示;20~30黄色;10~20绿色;0~10蓝色。使用点号'.'切换显示碱基和点号;使用r切换显示read name等还有很多其它的使用说明,具体按 ? 键来查看。
参考:samtools的说明文档:http://samtools.sourceforge.net/samtools.shtml
http://www.plob.org/2014/01/26/7112.html
查询需要根据前面建立的索引来做。
这是一个比较复杂的过程,我也是看了bowtie的作者的ppt才慢慢弄懂的,感觉自己也不可能三言两语就说清楚,一般都是辅助图片,动画,再经过多方交流才能慢慢理解。
所以大家呢,就自己去看ppt,看懂那个查询算法。(ppt及代码在我的群里面有共享,欢迎大家加群交流)
这里我简单讲讲我的程序
首先读取索引文件,统计好A,C,G,T的总数
然后把查询序列从最后一个字符往前面回溯。
我创建了一个子函数,专门来处理回溯的问题
每次接受四个参数(左右两端的碱基,上下的阈值),并返回两个参数(新的上下两个阈值)
大家要看懂阈值是如何更新迭代,这样动态的一个个回溯字符串,一个个迭代阈值。
直到四种临界情况的出现。
第一是上下阈值已经相等了,但是我们还没有回溯完全,那就说明字符串只能查找后几个字符,前面还有字符是无法匹配的
第二种情况是上下阈值已经相等了,正巧我们也回溯到了最后一个字符串,那么我们就找到了精确匹配。
第三种情况是已经进行到了最后一个字符串,但是上下阈值还有差值,那么就找到了多个精确匹配点。
最后一种情况是各种非法字符。
然后我简单的测序了一下在病毒的5K基因组里面的精确匹配情况,好像效果还挺好的
但是在酵母里面还有一个问题没有解决,就是取前二十个字符串排序的问题,不够精确,需要重新审视排序结果进行局部优化,可能是需要用堆排序发,具体我还得考虑一个星期,只能等下周上课再看看了,平时太忙了,基本没时间码代码。
这里贴上我的代码给大家看看,
[perl]
$a='CGCTATGTACTGGATGCGCTGGCAAACGAGCCTGCCGTAAG';
while(<>){
chomp;
@F=split;
$hash_count_atcg{$F[0]}++;
$hash{$.}=$_;
}
$all_a=$hash_count_atcg{'A'};
$all_c=$hash_count_atcg{'C'};
$all_g=$hash_count_atcg{'G'};
$all_t=$hash_count_atcg{'T'};
#print "$all_a\t$all_c\t$all_g\t$all_t\n";
$len_a=length $a;
$end_a=$len_a-1;
print "your query is $a\n";
print "and the length of your query is $len_a \n";
foreach (reverse (0..$end_a)){
$after=substr($a,$_,1);
$before=substr($a,$_-1,1);
#对第一个字符进行找阈值的时候,我们需要人为的定义起始点!
if($_ == $end_a){
if ($after eq 'A') {
$start=1;
$end=$all_a;
}
elsif ($after eq 'C') {
$start=$all_a+1;
$end=$all_a+$all_c;
}
elsif ($after eq 'G') {
$start=$all_a+$all_c+1;
$end=$all_a+$all_c+$all_g;
}
elsif ($after eq 'T'){
$start=$all_a+$all_c+$all_g+1;
$end=$all_a+$all_c+$all_g+$all_t;
}
else {print "error !!! we just need A T C G !!!\n";exit;}
}
#如果阈值已经无法继续分割,但是字符串还未查询完
if ($_ > 0 && $start == $end) {
$find_char=substr($a,$_);
$find_len=length $find_char;
#这里需要修改,但是不影响完全匹配了
print "we can just find the last $find_len char ,and it is $find_char \n";
exit;
}
#如果进行到了最后一个字符
if ($_ == 0) {
if ($start == $end) {
print "It is just one perfect match ! \n";
my @F_start=split/\s+/,$hash{$start};
print "The index is $F_start[1]\n";
exit;
}
else {
print "we find more than one perfect match!!!\n";
#print "$start\t$end\n";
foreach ($start..$end) {
my @F_start=split/\s+/,$hash{$_};
print "One of the index is $F_start[1]\n";
}
exit;
}
}
($start,$end)=&find_level($after,$before,$start,$end);
}
sub find_level{
my($after,$before,$start,$end)=@_;
my @F_start=split/\s+/,$hash{$start};
my @F_end=split/\s+/,$hash{$end};
if ($before eq 'A') {
return ($F_start[2],$F_end[2]);
}
elsif ($before eq 'C') {
return ($all_a+$F_start[3],$all_a+$F_end[3]);
}
elsif ($before eq 'G') {
return ($all_a+$all_c+$F_start[4],$all_a+$all_c+$F_end[4]);
}
elsif ($before eq 'T') {
return ($all_a+$all_c+$all_g+$F_start[5],$all_a+$all_c+$all_g+$F_end[5]);
}
else {print "sorry , I can't find the right match!!!\n";}
}
#perl -alne '{next if />/;$all.=$_;}END{print substr($all,308,10)}' lambda_virus.fa
[/perl]
其中第一讲我提到了一个简单的索引产生方式,因为是课堂就半个小时想的,很多细节没有考虑到,对病毒那种几K大小的基因组来说是很简单的,速度也非常快,但是我测试了一下酵母,却发现好几个小时都没有结果,我只好kill掉重新改写算法,我发现之前的测序最大的问题在于没有立即substr函数的实现方式,把一个5M的字符串不停的截取首尾字符串好像是一个非常慢的方式。
所以我优化了那个字符串的函数,虽然代码量变多了,实现过程也繁琐了一点,但是速度提升了几千倍。
time perl bwt_new_index.pl e-coli.fa >e-coli.index
测试了一下我的脚本,对酵母这样的5M的基因组,索引耗费时间是43秒
real 0m43.071s
user 0m41.277s
sys 0m1.779s
输出的index矩阵如下,我简单的截取头尾各10行给大家看,一点要看懂这个index。
首先第一列就是我们的BWT向量,也就是BWT变换后的尾字符
第二列是之前的顺序被BWT变换后的首字符排序后的打乱的顺序。
第三,四,五,六列分别是A,C,G,T的计数,就是在当行之前累积出现的A,C,G,T的数量,是对第一列的统计。
这个索引文件将会用于下一步的查询,这里贴上我新的索引代码,查询见下一篇文章
[perl]
while (<>){
next if />/;
chomp;
$a.=$_;
}
$len=length $a;
open FH_F,">tmp_forward.txt";
open FH_R,">tmp_reverse.txt";
for(my $i=0;$i<=$len-1;$i+=20){
print FH_F substr($a,$i,20);
print FH_F "\n";
}
$rev_a=reverse $a;
for(my $i=0;$i<=$len-1;$i+=20){
print FH_R substr($rev_a,$i,20);
print FH_R "\n";
}
close FH_F;
close FH_R;
$a='';
open FH_F,"tmp_forward.txt";
open FH_R,"tmp_reverse.txt";
#把前一行的所有20bp碱基当做后一行的头部信息
$residue_F=<FH_F>;
$residue_R=<FH_R>;
$i=0;
while ($F_reads=<FH_F>){
$R_reads=<FH_R>;
$F_merge=$residue_F.$F_reads;
$R_merge=$residue_R.$R_reads;
#这样每次就需要处理20个碱基
foreach (0..19) {
$up =substr($F_merge,$_,20);
$down=substr($R_merge,$_,1);
$hash{"$up\t$down"}=$i;
$i++;
}
#处理完毕之后再保存当行的20bp碱基做下一行的头部信息
$residue_F=$F_reads;
$residue_R=$R_reads;
}
#print "then we sort it\n";
$count_a=0;
$count_c=0;
$count_g=0;
$count_t=0;
foreach (sort keys %hash){
$first=substr($_,0,1);
$len=length;
$last=substr($_,$len-1,1);
#print "$first\t$last\t$hash{$_}\n";
$count_a++ if $last eq 'A';
$count_c++ if $last eq 'C';
$count_g++ if $last eq 'G';
$count_t++ if $last eq 'T';
print "$last\t$hash{$_}\t$count_a\t$count_c\t$count_g\t$count_t\n";
}
unlink("tmp_forward.txt");
unlink("tmp_reverse.txt");
[/perl]
一.阅读文献找到SRP
该文献讲了单分子测序在医疗领域的一个应用,我感觉挺重要的,就分析了一下,然后下载了数据,准备处理一下。
Single-step capture and sequencing of natural DNA for detection of BRCA1 mutations
在NCBI查到该数据地址,并且用脚本下载即可
http://www.ncbi.nlm.nih.gov/sra/?term=SRP007097
下载之后的数据如下,共19个测序文件,都是200K左右大小,那两个一百多M的可能是下载错了
for i in {32..52}
do
wget ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR258/SRR2588$i/SRR2588$i.sra
Done
下载的19个数据,都是只有1万多条序列。
因为这些判断都是对BRCA1这个基因进行目标性测序,所以接下来需要对它们进行特殊的处理。
感觉大家对很多生物信息学的术语都不甚了解,我这里简单的从一个基因开始,扩展开来讲一讲生信数据库,及它相关的一些术语!
我要讲的基因是BRCA1,这是一个与乳腺癌以及卵巢癌都息息相关的基因。而BRCA1是它的英文缩写简称,也是通常学者们进行交流十它的名字。它的全称是breast cancer 1,每个基因都会有一个简称,比如下面这些,在human里面这些简称多大47732个,正常人都不会认识它们所有,只需要碰到了去数据库搜索即可,但是搞医疗健康的,必须熟悉癌症50基因。
这样的缩写简称其实弊端很多,单词毕竟是有限的,而且缩写也没有语义。所以NCBI给每个基因都定义了一个entrez ID号,是整数的排序,具体大家可以去看NCBI发的一篇文献,专门讲解了entrez ID号的好处。
1 A1BG
2 A2M
3 A2MP1
9 NAT1
10 NAT2
11 NATP
12 SERPINA3
13 AADAC
14 AAMP
这里我们来找一下我们的BRCA1这个基因在生物信息数据库里面的其它信息,在NCBI的ftp里面有一个文件是Homo_sapiens.gene_info里面包含着人类所以基因的全部信息
9606 首先这个基因在human上面的,而human被NCBI定义的taxid是9606
672 然后这个基因的被NCBI定义的entrez ID号是672
BRCA1 这个当然就是这个基因的英文缩写名称啦
- 这个表明这个基因在负链什么
BRCAI|BRCC1|BROVCA1|FANCS|IRIS|PNCA4|PPP1R53|PSCP|RNF53
这个可能是基因以前的名称,或者是在其它研究领域的一些名称。MIM:113705|HGNC:HGNC:1100|Ensembl:ENSG00000012048|HPRD:00218|Vega:OTTHUMG00000157426
这里面包含在它在其它数据库的信息,我们的NCBI用entrez ID号672来标识它,相应的ensembl数据用ensembl ID号ENSG00000012048来标识它,还有什么MIM数据库,HGNC数据库,Vega数据库我就不详细讲啦
17 17q21 这个说明它在human的17号染色体的位置信息
下面一堆都是这个基因的描述,它的功能等等。
breast cancer 1, early onset protein-coding BRCA1 breast cancer 1, early onset
O BRCA1/BRCA2-containing complex, subunit 1|Fanconi anemia, complementation group S|RING finger protein 53|breast and ovarian cancer susceptibility protein 1|breast and ovarian cancer sususceptibility protein 1|breast cancer type 1 susceptibility protein|protein phosphatase 1, regulatory subunit 53
20150201
这样我们就把好几个数据库给串起来了,也大致了解了一个基因的各种信息,但是,这样肯定是不够的。
接下来我们就不用BRCA1来称呼这个基因了,我们统一用NCBI定义entrez ID号672来称呼这个基因,当然用ensembl ID号ENSG00000012048也可以,它们都是比较通用的。
ENSG00000012048 672 这个基因在GO数据库里面可以找到67个功能信息,分别是以下
GO:0000151 GO:0000724 GO:0000724 GO:0000794 GO:0003677 GO:0003684 GO:0003713 GO:0003723 GO:0004842 GO:0005515 GO:0005634 GO:0005654 GO:0005694 GO:0005737 GO:0005886 GO:0006260 GO:0006281 GO:0006301 GO:0006302 GO:0006302 GO:0006349 GO:0006357 GO:0006359 GO:0006633 GO:0006915 GO:0006974 GO:0006978 GO:0007059 GO:0007098 GO:0008270 GO:0008274 GO:0008630 GO:0009048 GO:0010212 GO:0010575 GO:0010628 GO:0015631 GO:0016567 GO:0016874 GO:0019899 GO:0030521 GO:0030529 GO:0031398 GO:0031436 GO:0031572 GO:0031625 GO:0035066 GO:0035067 GO:0042127 GO:0042981 GO:0043009 GO:0043234 GO:0043627 GO:0044030 GO:0044212 GO:0045717 GO:0045739 GO:0045766 GO:0045892 GO:0045893 GO:0045893 GO:0045944 GO:0045944 GO:0046600 GO:0050681 GO:0051571 GO:0051572 GO:0051573 GO:0051574 GO:0051865 GO:0070512 GO:0070531 GO:0071158 GO:0071356 GO:0071681 GO:0085020 GO:1902042 GO:2000378 GO:2000617 GO:2000620
由于GO太多了,我简单讲几个
ubiquitin ligase complex
double-strand break repair via homologous recombination
double-strand break repair via homologous recombination
condensed nuclear chromosome
DNA binding
damaged DNA binding
transcription coactivator activity
RNA binding
ubiquitin-protein transferase activity
protein binding
都是描述这个基因的功能的。
到这里我们大致了解了这个基因的功能,但是还不够。
然后可以查到它有一下6个转录本,都有二十多个外显子。
NR_027676
NM_007300
NM_007299
NM_007298
NM_007297
NM_007294
在hg19这个参考基因组的起始终止坐标,还有各个外显子的起始终止坐标都能找到。
41196311,41199659,41201137,41203079,41209068,41215349,41215890,41219624,41222944,41226347,41228504,41234420,41242960,41243451,41247862,41249260,41251791,41256138,41256884,41258494,41267742,41276033,41277198
41197819,41199720,41201211,41203134,41209152,41215390,41215968,41219712,41223255,41226538,41228631,41234592,41243049,41246877,41247939,41249306,41251894,41256278,41256973,41258550,41267796,41276132,41277340
http://asia.ensembl.org/Homo_sapiens/Gene/Summary?db=core;g=ENSG00000012048;r=17:43044295-43125483
在ensembl里面关于这个基因的描述如下。
breast cancer 1, early onset [Source:HGNC Symbol;Acc:HGNC:1100]
BRCC1, FANCS, PPP1R53, RNF53
Chromosome 17: 43,044,295-43,125,483 reverse strand.
chromosome:GRCh38:CM000679.2:43044295:43125483:1
This gene has 29 transcripts (splice variants), 63 orthologues, is a member of 4 Ensembl protein families and is associated with 11 phenotypes.
RefSeq Gene ID 672
Uniprot identifiers: P38398
而且ensembl里面可以可视化这个基因的所有信息。
然后简单检索一下关于这个BRCA1基因的文献发表状况,居然多达2111篇文献,看来这个基因很火呀!!!
awk '{if ($1==9606 && $2==672) print }' gene2pubmed |wc
9606 672 1676470
9606 672 2001833
9606 672 2270482
9606 672 4506230
9606 672 7481765
9606 672 7545954
9606 672 7550349
9606 672 7795652
9606 672 7894491
9606 672 7894492
第三列1676470等编号是pubmed数据库的文献编号,可以直接找到关于这个基因的文献发表情况。
而直接在NCBI的pubmed数据库里面可以搜到多达11339篇文献。
esearch -db pubmed -query 'BRCA1'
Esearch这个程序是NCBI提供的,挺好用的,希望大家可以熟悉一下。
esearch -db pubmed -query 'BRCA1' | efetch -format docsum | xtract -pattern DocumentSummary -present Author -and Title -element Id -first "Author/Name" -element Title >BRCA1.pubmed
用这个代码,可以找到所有关于这个BRCA1基因的文献的作者及标题,这样可以统计在这个基因领域的研究者最出名的是谁。
至于这个基因的序列,及其转录本翻译的蛋白我就不列了,太长了,而且占位子
这里我选取酵母基因组来组装,以为它只有一条染色体,而且本身也不大!
这个文件就4.5M,然后第一行就是序列名,第二列就是序列的碱基组成。共4641652个碱基。
我写一个perl程序来人为的创造一个测序文件
这样我们的4.5M基因组就模拟出来了486M的单端100bp的测序数据,而且是无缝连接,按照道理应该很容易就拼接的。
/home/jmzeng/bio-soft/SOAPdenovo2-bin-LINUX-generic-r240/SOAPdenovo-127mer
all -s config_file -K 63 -R -o graph_prefix 1>ass.log 2>ass.err
可以看到组装效果还不错哦,然后我模拟了一个测试数据,再进行组装一次,这次更好!
其实还可以模拟双端测序,应该就能达到百分百组装了。
但是由于我代码里面选取的是80在随机错开,所以我把kmer的长度设置成了81来试试看,希望这样可以把它完全组装成一条e-coli基因组。
/home/jmzeng/bio-soft/SOAPdenovo2-bin-LINUX-generic-r240/SOAPdenovo-127mer
all -s config_file -K 81 -R -o graph_prefix 1>ass.log 2>ass.err
但是也没有什么实质性的提高,虽然理论上是肯定可以组装到一起!
那我再模拟一个双端测序吧,中间间隔200bp的。
一.下载并安装这个软件
下载地址进下面,但是下载源码安装总是很困难,我直接下载bin文件可执行程序。
解压进入目录
首先make
然后make install即可
安装总是失败,我也不知道怎么回事,懒得解决了。
直接去我老师那里把这个程序拷贝进来了。
https://github.com/aquaskyline/SOAPdenovo2/archive/master.zip
http://sourceforge.net/projects/soapdenovo2/files/latest/download?source=files
也可以直接下载bin程序
二.准备测试数据
类似于这样的几个文库的左右两端测序数据。
我这里用一个小样本的单端数据做测试
三,参考命令
You may run it like this:
参考:http://www.plob.org/2012/07/06/2537.html
https://github.com/aquaskyline/SOAPdenovo2
总共就四个步骤,介绍如下。
| ./pregraph_sparse [parameters] |
| ./SOAPdenovo-63mer contig [parameters] |
| ./SOAPdenovo-63mer map [parameters] |
| ./SOAPdenovo-63mer scaff [parameters] |
| i) preparing the pregraph. This step is similar to velveth for velvet. |
| ii) Determining contigs. This step is similar to velvetg for velvet. |
| iii) Mapping back reads on to contigs. |
| iv) Assembling contigs into scaffolds. |
| SOAPdenovo-63mer sparse_pregraph -s config_file -K 45 -p 28 -z 1100000000 -o outPG |
| SOAPdenovo-63mer contig -g outPG |
| SOAPdenovo-63mer map -s config_file -g outPG -p 28 |
| SOAPdenovo-63mer scaff -g outPG -p 28 |
官网给出的步骤如下
这个命令还需要一个配置文件
max_rd_len=99 设置最大reads长度,具体情况具体定义
[LIB] 第一个文库数据
avg_ins=225
reverse_seq=0
asm_flags=3
rank=1
q1=runPE_1.fq
q2=runPE_2.fq
[LIB] 第二个文库数据
avg_ins=2000
reverse_seq=1
asm_flags=2
rank=2
q1=runMP_1.fq
q2=runMP_2.fq
也可以全部一次性的搞一个命令
all -s config_file -K 63 -R -o graph_prefix 1>ass.log 2>ass.err
我简单修改了一下参考博客的代码跟官网的代码,然后运行了我自己的代码
/home/jmzeng/bio-soft/SOAPdenovo2-bin-LINUX-generic-r240/SOAPdenovo-127mer
all -s config_file -K 63 -R -o graph_prefix 1>ass.log 2>ass.err
反正我也不懂,就先跑跑看咯
我选取的是7个单端数据,所以我的配置文件是
max_rd_len=500
[LIB]
avg_ins=225
reverse_seq=0
asm_flags=3
rank=1
p=SRR072005.fa
p=SRR072010.fa
p=SRR072011.fa
p=SRR072012.fa
p=SRR072013.fa
p=SRR072014.fa
p=SRR072029.fa
四.输出数据解读
好像我的数据都比较小,就7个三百多兆的fasta序列,几个小时就跑完啦
四个步骤都有输出数据
好像组装效果惨不忍睹呀!共86万的contig,50多万的scaffold
scaffolds>100 505473 99.60%
scaffolds>500 113523 22.37%
scaffolds>1K 48283 9.51%
scaffolds>10K 0 0.00%
scaffolds>100K 0 0.00%
scaffolds>1M 0 0.00%
这其实都相当于没有组装了,因为我的测序判断本来就很多是大于500的!
可能是我的kmer值选取的不对
Kmer为63跑出来的效果不怎么好,86万的contig,50万的scaffold的
Kmer为35跑出来的效果更惨,203万的contig,近60万的scaffold。
我觉得问题可能不是这里了,可能是没有用到那个20k和3k的双端测序库,唉,其实我习惯了illumina的测序数据,不太喜欢这个454的
感觉组装好难呀,业余时间搞不定呀,希望有高手能一起交流,哈哈,我自己再慢慢来试试。
今天先 对7个单端数据做处理,是454数据,平均长度300bp左右,明天再处理3KB和20KB的配对reads。
首先跑fastqc
打开一个个看结果
可以看到前面一些碱基的质量还是不错的, 因为这是454平台测序数据,序列片段长度差异很大,一般前四百个bp的碱基质量还是不错的,太长了的测序片段也不可靠
重点在下面这个图片,可以看到,前面的4个碱基是adaptor,肯定是要去除的,不是我们的测序数据。是TCAG,需要去除掉。
所以我们用了 solexaQA 这个套装软件对原始测序数据进行过滤
可以看到过滤的非常明显!!!甚至有个样本基本全军覆没了!然后我查看了我的批处理脚本,发现可能是perl DynamicTrim.pl -454 $id这个参数有问题
for id in *fastq
do
echo $id
perl DynamicTrim.pl -454 $id
done
for id in *trimmed
do
echo $id
perl LengthSort.pl $id
done
可以看到末尾的质量差的碱基都被去掉了,但是头部的TCAG还是没有去掉。
处理完毕后的数据如下:
一.下载该软件
http://solexaqa.sourceforge.net/index.htm
下载解压开
现在已经把它的三个功能整合到一起啦
之前是分开的程序,我主要用它的两个perl 程序,我比较喜欢之前的版本,所以下面的讲解也是基于这两个perl程序。
这两
个主要是对reads进行最大子串的截取
二.准备数据。
就是我们测序得到的原始数据。
第一个就是质量控制,一般是以20为标准,当然你也可以自己设定,该软件质控的原理如下:
使用默认的参数值(defaults to P = 0.05, or equivalently, Q = 13)
基本上就是取符合阈值的最大子串。
二:命令使用很简单一般使用DynamicTrim与LengthSort.pl就可以了
for id in *fastq
do
echo $id
perl DynamicTrim.pl -454 $id
done
for id in *trimmed
do
echo $id
perl LengthSort.pl $id
done
首先使用DynamicTrim.pl程序,非常耗时间
几个小时完毕之后
查看,产出文件如下
可以看到丢弃的不多,也就三五百M的
简单查看丢弃的,都是短的。
perl -lne '{print length if $.%4==2}' SRR1793918.fastq.trimmed.discard |head
用这个脚本查看,可知好像都是短于25个碱基的被舍弃掉了,这个参数可以调整的。
接下来就可以用这些数据进行数据分析了
其实我现在一般都用的是blast++了,也专门写了篇日志介绍它!
但是看到一些就的服务器上面只有blast,所以就搜了一些它的用法。
主要参考 http://www.bio.ku.dk/nuf/resources/BLAST_index.htm
很简单的两个步骤
首先建库formatdb -i Cad16_aa.fasta -p T -o F
就是把 Cad16_aa.fasta这个序列文件变成blast专用的库,-p选项中的T是代表蛋白库
然后就比对咯,比对程序有六个,需要用-p来选择
blastall -p blastx -d nr -i 19A.fa -o 19A.outm -v 1 -b 1 -m 8
上面这个命令就是选择了blastx这个比对程序,数据库是nr ,输入的查询序列是 19A.fa
然后我们输出格式的m8,这个格式很重要,我们还可以设置-a控制cpu数量,和-e控制阈值
|
BLAST programs |
|
| blastp | Protein query > Protein database |
| blastn | Nucleotide query > Nucleotide database |
| blastx | Nucleotide query > Protein database (via translated query) |
| tblastn | Protein query > Nucleotide database (via translated database) |
| tblastx | Nucleotide query > Nucleotide database (via translated query and database) |
|
Formatting database for local BLAST |
|
| - | Show a list of all arguments. |
| -i | Input file(s) for formatting. Optional. |
| -p | Type of file [T/F]. T = protein, F = nucleotide. Default = T. |
| -o | Parse option [T/F]. T = Parse SeqId and create indexes, F = Do not parse or create indexes. |
http://statgenpro.psychiatry.hku.hk/limx/kggseq/download/resources/
这个网站收集了大部分资料,我们就用它的,如果它倒闭了,大家再想办法去搜索吧。
其实这些文件都是基于NCBI以及UCSC和ensembl数据库的文件用一些脚本转换而来的,都是非常简单的脚本。
首先我们看看humandb/hg19_refGene.txt 这个文件,总共2.5万多个基因的共5万多个转录本。
19 可能是entrez ID,但是又不像。
NM_001291929 参考基因名
chr11 染色体
-
89057521
89223909
89059923
89223852
17 89057521,89069012,89070614,89073230,89075241,89088129,89106599,89133184,89133382,89135493,89155069,89165951,89173855,89177302,89182607,89184952,89223774, 89060044,89069113,89070683,89073339,89075361,89088211,89106660,89133247,89133547,89135710,89155150,89166024,89173883,89177400,89182692,89185063,89223909,
0
NOX4 基因的英文简称,通俗名
cmpl
cmpl
2,0,0,2,2,1,0,0,0,2,2,1,0,1,0,0,0,
然后我们看看hg19_snp141.txt这个文件
1 10229 A - .
1 10229 AACCCCTAACCCTAACCCTAAACCCTA - .
1 10231 C A .
1 10231 C - .
1 10234 C T .
1 10248 A T .
1 10250 A C .
1 10250 AC - .
1 10255 A - .
1 10257 A C .
1 10259 C A .
1 10291 C T .
1 10327 T C .
1 10329 ACCCCTAACCCTAACCCTAACCCT - .
1 10330 C - .
1 10390 C - .
1 10440 C A .
1 10440 C - .
1 10469 C G .
1 10492 C T .
1 10493 C A .
1 10519 G C .
1 10583 G A 0.144169
1 10603 G A .
1 10611 C G 0.0188246
1 10617 CGCCGTTGCAAAGGCGCGCCG -
里面记录了以hg19为参考的所有的snp位点。
585
ENST00000518655 基因的ensembl ID号
chr1 + 11873 14409 14409 14409
4 基因有四个外显子
11873,12594,13402,13660, 12227,12721,13655,14409, 在基因的四个外显子的坐标
0
DDX11L1 基因的通俗英文名
none none -1,-1,-1,-1,
CTTGCCGTCAGCCTTTTCTTT·····gene的核苷酸序列
一、下载及安装软件
这个软件需要edu邮箱注册才能下载,可能是仅对科研高校开放吧。所以软件地址我就不列了。
它其实是几个perl程序,比较重要的是这个人类的数据库,snp注释必须的。
参考:http://annovar.readthedocs.org/en/latest/misc/accessory/
二,准备数据
既然是注释,那当然要有数据库啦!数据库倒是有下载地址
http://www.openbioinformatics.org/annovar/download/hg19_ALL.sites.2010_11.txt.gz
也可以用命令来下载
Perl ./annotate_variation.pl -downdb -buildver hg19 -webfrom annovar refGene humandb/
然后我们是对snp-calling流程跑出来的VCF文件进行注释,所以必须要有自己的VCF文件,VCF格式详解见本博客另一篇文章,或者搜索也行
http://vcftools.sourceforge.net/man_latest.html
三、运行的命令
首先把vcf格式文件,转换成空格分隔格式文件,自己写脚本也很好弄
perl convert2annovar.pl -format vcf
/home/jmzeng/raw-reads/whole-exon/snp-calling/tmp1.vcf >annovar.input
变成了空格分隔的文件
然后把转换好的数据进行注释即可
./annotate_variation.pl -out ex1 -build hg19 example/ex1.avinput humandb/
四,输出文件解读
比对可以选择BWA或者bowtie,测序数据可以是单端也可以是双端,我这里简单讲一个,但是脚本都列出来了。而且我选择的是bowtie比对,然后单端数据。
首先进入hg19的目录,对它进行两个索引
samtools faidx hg19.fa
Bowtie2-build hg19.fa hg19
我这里随便从26G的测序数据里面选取了前1000行做了一个tmp.fa文件,进入tmp.fa这个文件的目录进行操作
Bowtie的使用方法详解见http://www.bio-info-trainee.com/?p=398
bowtie2 -x ../../../ref-database/hg19 -U tmp1.fa -S tmp1.sam
samtools view -bS tmp1.sam > tmp1.bam
samtools sort tmp1.bam tmp1.sorted
samtools index tmp1.sorted.bam
samtools mpileup -d 1000 -gSDf ../../../ref-database/hg19.fa tmp1.sorted.bam |bcftools view -cvNg - >tmp1.vcf
然后就能看到我们产生的vcf变异格式文件啦!
当然,我们可能还需要对VCF文件进行再注释!
要看懂以上流程及命令,需要掌握BWA,bowtie,samtools,bcftools,
数据格式fasta,fastq,sam,vcf,pileup
如果是bwa把参考基因组索引化,然后aln得到后缀树,然后sampe对双端数据进行比对
首先bwa index 然后选择算法,进行索引。
然后aln脚本批量处理
==> bwa_aln.sh <==
while read id
do
echo $id
bwa aln hg19.fa $id >$id.sai
done <$1
然后sampe脚本批量处理
==> bwa_sampe.sh <==
while read id
do
echo $id
bwa sampe hg19.fa $id*sai $id*single >$id.sam
done <$1
然后是samtools的脚本
==> samtools.sh <==
while read id
do
echo $id
samtools view -bS $id.sam > $id.bam
samtools sort $id.bam $id.sorted
samtools index $id.sorted.bam
done <$1
然后是bcftools的脚本
==> bcftools.sh <==
while read id
do
echo $id
samtools mpileup -d 1000 -gSDf ref.fa $id*sorted.bam |bcftools view -cvNg - >$id.vcf
done <$1
==> mpileup.sh <==
while read id
do
echo $id
samtools mpileup -d 100000 -f hg19.fa $id*sorted.bam >$id.mpileup
done <$1
需要插件和自己修改主题下面的foot.php代码。
参考 http://jingyan.baidu.com/article/ae97a646ce37c2bbfd461d01.html
步骤如下:
1、登陆到wp后台,鼠标移动到左侧菜单的“插件”链接上,会弹出子菜单,点击子菜单的“安装插件”链接
2、WP-PostViews插件显示wordpress文章点击浏览量
在“安装插件”链接页面的搜索框中输入“WP-PostViews”,然后回车
3、WP-PostViews插件显示wordpress文章点击浏览量
在搜索结果页面点击“WP-PostViews”插件内容区域的“现在安装”按钮
4、WP-PostViews插件显示wordpress文章点击浏览量
程序自动下载插件到服务器并解压安装,一直等到安装成功信息出现,然后在安装成功提示页面点击“启动插件”链接。
5、WP-PostViews插件显示wordpress文章点击浏览量
页面会自动跳转到“已安装插件”页面,在已安装插件列表中我们可以看到“Form Manager”插件已经处于启用状态(插件名下是“停用”链接)。
有了这个插件之后,我们的整个网页环境里面就多了一个 the_views()函数,它统计着每个文章的点击数,这样我们之前的网页就能显示点击数了。
这个是我现在用的主题的php代码,把文章用span标记隔开了,而且显示着上面php代码里面的每一个内容包括日期,分类,标签,评论等等
其中thez-view()这个函数返回的不仅仅是一个访客数,但是我的文章的访客都太少了,所以我写了一个脚本帮我刷一刷流量。
[perl]
use List::MoreUtils qw(uniq);
$page='http://www.bio-info-trainee.com/?paged=';
foreach (1..5){ #我的文章比较少,就42个,所以只有5个页面
$url_page=$page.$_;
$tmp=`curl $url_page`;
#@p=$tmp=~/p=(\d+)/;
$tmp =~ s/(p=\d+)/push @p, $1/eg; #寻找p=数字这样的标签组合成新的网页地址
}
@p=uniq @p;
print "$_\n" foreach @p; #可以找到所有42个网页的地址
foreach (@p){
$new_url='http://www.bio-info-trainee.com/?'.$_;
`curl $new_url` foreach (1..100); #每个网页刷一百次
}
[/perl]
大家可以看到这个网页被刷的过程,从15到21到27直到100
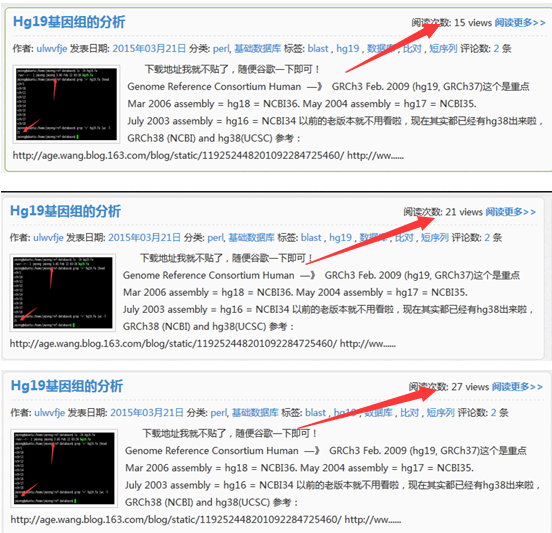
大家现在再去看我的网页,就每个文章都有一百的访问量啦!
http://www.bio-info-trainee.com/