这一讲其实算不上是自学miRNA-seq分析,本质就是affymetrix的mRNA表达芯片数据分析,而且还是最常用的那种GPL570 HG-U133_Plus_2,但是因为是跟miRNA样本配对检测的,而且后面会利用到这两个数据分析结果来做共表达网络分析等等,所以就贴出对该芯片数据的分析结果。文章里面也提到了 Messenger RNA expression analysis identified 731 probe sets with significant differential expression,作者挑选的差异分析结果的显著基因列表如下: Continue reading
Tag Archives: 差异分析
自学miRNA-seq分析第六讲~miRNA表达量差异分析
这一讲是miRNA-seq数据分析的分水岭,前面的5讲说的是读文献下载数据比对然后计算表达量,属于常规的流程分析,一般在公司测序之后都可以拿到分析结果,或者文献也会给出下载结果。但是单纯的分析一个样本意义不大,一般来说,我们做研究都是针对于不同状态下的miRNA表达量差异分析,然后做注释,功能分析,网络分析,这才是重点,也是难点。我这里就直接拿文献处理好的miRNA表达量来展示如何做下游分析,首先就是差异分析啦: Continue reading
用samr包对芯片数据做差异分析
本来搞差异分析的工具和包就一大堆了,而且limma那个包已经非常完善了,我是不准备再讲这个的,正好有个同学问了一下这个包,我就随手测试了一下,顺便看看它跟limma有什么差异没有!手痒了就记录了测试流程!
学习一个包其实非常简单,就是找到包的官网看看说明书即可!说明书链接
用limma包的voom函数来对RNA-seq数据做差异分析
limma真不愧是最流行的差异分析包,十多年过去了,一直是芯片数据处理的好帮手。
现在又可以支持RNA-seq数据,我赶紧试用了一下!
我下面只讲用法,大家看代码就明白了!
用R语言的DESeq2包来对RNA-seq数据做差异分析
我以前写过DESeq,以及过时了:http://www.bio-info-trainee.com/867.html
正好准备筹集bioconductor中文社区,我写简单讲一下DESeq2这个包如何用!
差异分析是否需要比较矩阵
最流行的差异分析软件就是limma了,它现在更新了一个voom的算法,所以既可以对芯片数据,也可以对转录组高通量测序数据进行分析,其它所有的差异分析软件其实都是模仿这个的。
我以前讲到过做差异分析,需要三个数据:
- 表达矩阵
- 分组矩阵
- 差异比较矩阵
前面两个肯定是必须的,有表达矩阵,样本必须进行分组,才能分析,但是我看到过好几种例子,有的有差异比较矩阵,有的没有。
后来我仔细研究了一下limma包的说明书,发现这其实是一个很简单的问题。
大家仔细观察下面的两个代码
首先是不需要差异比较矩阵的
library(CLL)
data(sCLLex)
library(limma)
design=model.matrix(~factor(sCLLex$Disease))
fit=lmFit(sCLLex,design)
fit=eBayes(fit)
options(digits = 4)
#topTable(fit,coef=2,adjust='BH')
> topTable(fit,coef=2,adjust='BH')
logFC AveExpr t P.Value adj.P.Val B
39400_at 1.0285 5.621 5.836 8.341e-06 0.03344 3.234
36131_at -0.9888 9.954 -5.772 9.668e-06 0.03344 3.117
33791_at -1.8302 6.951 -5.736 1.049e-05 0.03344 3.052
1303_at 1.3836 4.463 5.732 1.060e-05 0.03344 3.044
36122_at -0.7801 7.260 -5.141 4.206e-05 0.10619 1.935
36939_at -2.5472 6.915 -5.038 5.362e-05 0.11283 1.737
41398_at 0.5187 7.602 4.879 7.824e-05 0.11520 1.428
32599_at 0.8544 5.746 4.859 8.207e-05 0.11520 1.389
36129_at 0.9161 8.209 4.859 8.212e-05 0.11520 1.389
37636_at -1.6868 5.697 -4.804 9.355e-05 0.11811 1.282
然后是需要差异比较矩阵的
library(CLL)
data(sCLLex)
library(limma)
design=model.matrix(~0+factor(sCLLex$Disease))
colnames(design)=c('progres','stable')
fit=lmFit(sCLLex,design)
cont.matrix=makeContrasts('progres-stable',levels = design)
fit2=contrasts.fit(fit,cont.matrix)
fit2=eBayes(fit2)
options(digits = 4)
topTable(fit2,adjust='BH')
logFC AveExpr t P.Value adj.P.Val B
39400_at -1.0285 5.621 -5.836 8.341e-06 0.03344 3.234
36131_at 0.9888 9.954 5.772 9.668e-06 0.03344 3.117
33791_at 1.8302 6.951 5.736 1.049e-05 0.03344 3.052
1303_at -1.3836 4.463 -5.732 1.060e-05 0.03344 3.044
36122_at 0.7801 7.260 5.141 4.206e-05 0.10619 1.935
36939_at 2.5472 6.915 5.038 5.362e-05 0.11283 1.737
41398_at -0.5187 7.602 -4.879 7.824e-05 0.11520 1.428
32599_at -0.8544 5.746 -4.859 8.207e-05 0.11520 1.389
36129_at -0.9161 8.209 -4.859 8.212e-05 0.11520 1.389
37636_at 1.6868 5.697 4.804 9.355e-05 0.11811 1.282
大家运行一下这些代码就知道,两者结果是一模一样的。
而差异比较矩阵的需要与否,主要看分组矩阵如何制作的!
design=model.matrix(~factor(sCLLex$Disease))
design=model.matrix(~0+factor(sCLLex$Disease))
有本质的区别!!!
前面那种方法已经把需要比较的组做出到了一列,需要比较多次,就有多少列,第一列是截距不需要考虑,第二列开始往后用coef这个参数可以把差异分析结果一个个提取出来。
而后面那种方法,仅仅是分组而已,组之间需要如何比较,需要自己再制作差异比较矩阵,通过makeContrasts函数来控制如何比较!
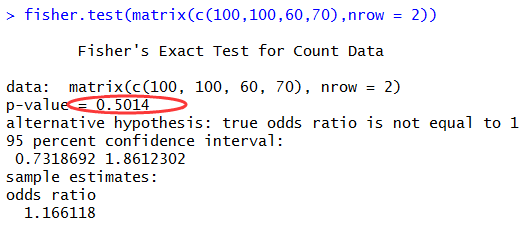
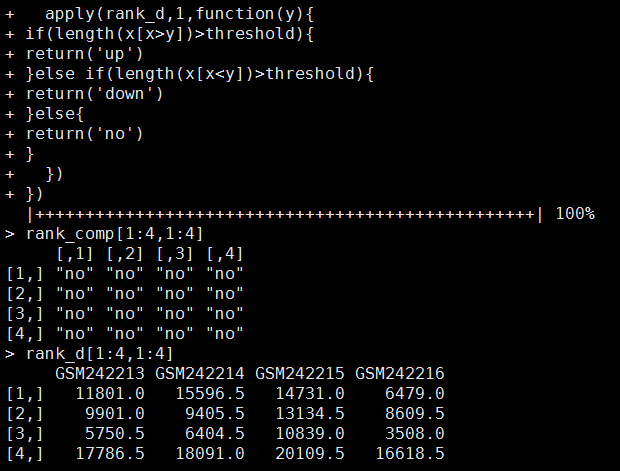
用RankComp的思想来做差异基因分析
我现在还不是很确定这个方法,只是试一试,欢迎与我交流对该方法的讨论!
Wang H, Sun Q, Zhao W, et al. Individual-level analysis of differential expression of genes and pathways for personalized medicine[J]. Bioinformatics, 2014: btu522.
他们把它写成了一个R包,可以下载使用,但是必须用R2.15.2版本,我用了一下,不好用!
We can download the R code for in http://bioinformatics.oxfordjournals.org/content/31/1/62/suppl/DC1
他们这个程序真心不好用,但是很容易看懂算法,可以自己用R语言写一个来实现同样的过程!
关于limma包差异分析结果的logFC解释
首先,我们要明白,limma接受的输入参数就是一个表达矩阵,而且是log后的表达矩阵(以2为底)。
那么最后计算得到的logFC这一列的值,其实就是输入的表达矩阵中case一组的平均表达量减去control一组的平均表达量的值,那么就会有正负之分,代表了case相当于control组来说,该基因是上调还是下调。
我之前总是有疑问,明明是case一组的平均表达量和control一组的平均表达量差值呀,跟log foldchange没有什么关系呀。
后来,我终于想通了,因为我们输入的是log后的表达矩阵,那么case一组的平均表达量和control一组的平均表达量都是log了的,那么它们的差值其实就是log的foldchange
首先,我们要理解foldchange的意义,如果case是平均表达量是8,control是2,那么foldchange就是4,logFC就是2咯
那么在limma包里面,输入的时候case的平均表达量被log后是3,control是1,那么差值是2,就是说logFC就是2。
这不是巧合,只是一个很简单的数学公式log(x/y)=log(x)-log(y)
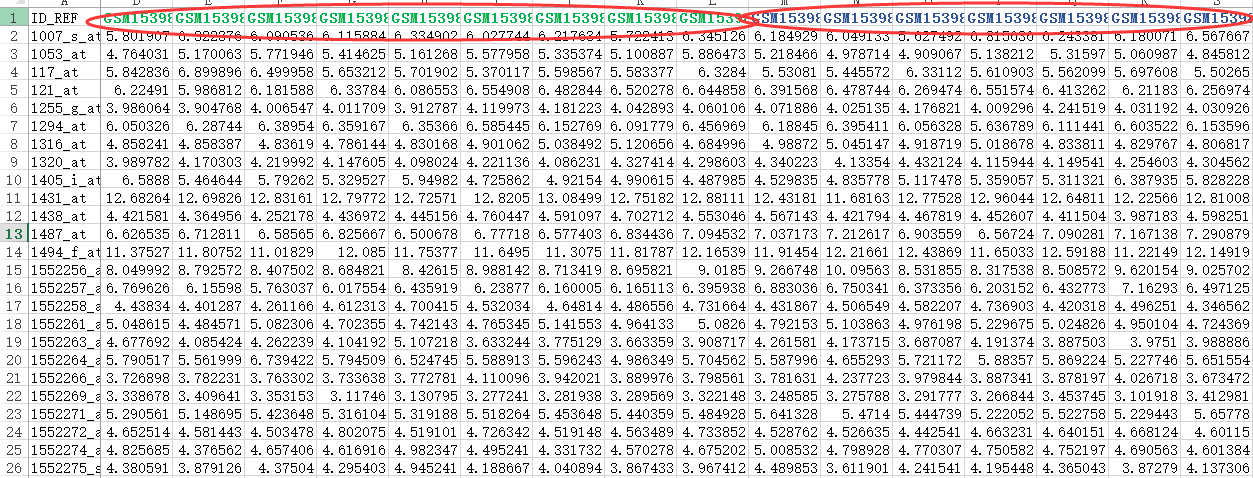
用excel表格做差异分析
其实主要要讲的不是用excel来做差异分析,只是想讲清楚差异分析的原理,用excel可视化的操作可能会更方便理解,而且想告诉大家,其实生物信息学分析,本来就很简单的,那么多软件,只有你理解了原理,你自己就能写出来的!
首先,还是得到表达矩阵,下面绿色的样本是NASH组,蓝色的样本是normal组
我们进行差异分析,很简单,就是看两组的表达值,是否差异,而检验的方法就是T检验。
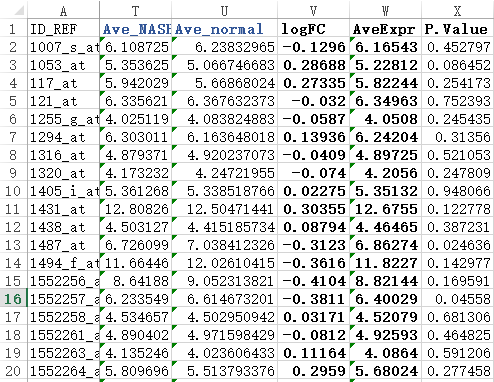
=AVERAGE(D2:L2) ##求NASH组的平均表达量
=AVERAGE(M2:S2) ###求normal的平均表达量
=T2-U2 ##计算得到logFOLDchange值
=AVERAGE(D2:S2) ###得到所有样本的平均表达量
=T.TEST(D2:L2,M2:T2,2,3) ###用T检验得到两个组的表达量的差异显著程度。
简单检查几个值就可以看到跟limma包得到的结果差不多。
用limma包对芯片数据做差异分析
下载该R语言包,然后看说明书,需要自己做好三个数据(表达矩阵,分组矩阵,差异比较矩阵),总共三个步骤(lmFit,eBayes,topTable)就可以啦
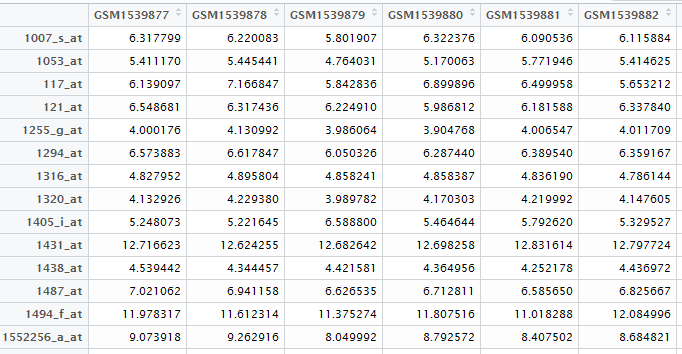
首先做第一个数据,基因表达矩阵!
自己在NCBI里面可以查到下载地址,然后用R语言读取即可
exprSet=read.table("GSE63067_series_matrix.txt.gz",comment.char = "!",stringsAsFactors=F,header=T)
rownames(exprSet)=exprSet[,1]
exprSet=exprSet[,-1]
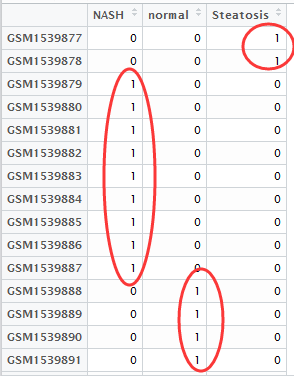
然后做好分组矩阵,如下
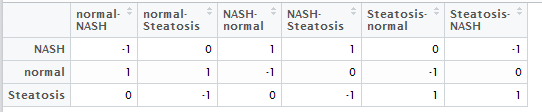
然后做好,差异比较矩阵,就是说明你想把那些组拿起来做差异分析,如下
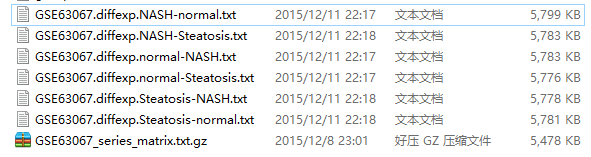
最后输出结果:
我进行了6次比较,所以会输出6次比较结果
最后打开差异结果,解读,说明书如下!
在我的github有完整代码