Tag Archives: 数据
【直播】我的基因组(九):拿到数据后要做的事情
时隔好几个月,因为各种各样的原因数据终于拿到了自己的手上,真是不容易啊!
拿到数据后,第一件要做的事情就是检查数据传输的完整性,然后备份!我拿到的数据如下:
可以看到,公司给了我测序仪的下机数据(raw data)和他们质控后的clean data,这个过程减少了6G的数据量,对应着约90亿bp的碱基,相当于减少了3个人的全基因组数据。具体推算公式见前面的系列直播贴!
首先我把数据拷贝到了我上上周买的2T移动硬盘里面,再拷贝到我工作电脑一份,服务器一份,私人电脑一份,另外一个移动硬盘一份。然后删除了公司寄给我的硬盘里面的数据,再把硬盘寄回给公司,然后监督他们删除我所有的数据。(做这么多就是为了保护隐私,当然这个大前提是我已经确定数据没有问题了。)
检查数据传输的完整性就是md5校验,看看数据在拷贝过程中有没有意外的损坏(这个在之前下载数据的时候我也说过)!一般传输数据之前,会用md5命令来生成各个文件的md5值,就是下面的MD5.txt文件里面的内容,然后传输数据之后,需要自行用md5sum -c MD5.txt 来校验文件里面记录的文件的完整性,如果显示都是OK,说明文件拷贝传输过程是没有问题的!但这个过程会耗费大量的磁盘读写,磁盘读写能力是有限的,所以开多个进程并不能加快这一过程。
然后我把公司处理好的bam文件上传到服务器做下游分析,我用的winscp软件把文件传到服务器上的!
从明天起,我们就开始正式对基因组进行分析啦!欢迎围观!
请扫描以下二维码关注我们,获取直播系列的所有帖子!
solexaQA 对测序数据进行简单过滤
一.下载该软件
http://solexaqa.sourceforge.net/index.htm
下载解压开
现在已经把它的三个功能整合到一起啦
之前是分开的程序,我主要用它的两个perl 程序,我比较喜欢之前的版本,所以下面的讲解也是基于这两个perl程序。
这两
个主要是对reads进行最大子串的截取
二.准备数据。
就是我们测序得到的原始数据。
第一个就是质量控制,一般是以20为标准,当然你也可以自己设定,该软件质控的原理如下:
使用默认的参数值(defaults to P = 0.05, or equivalently, Q = 13)
基本上就是取符合阈值的最大子串。
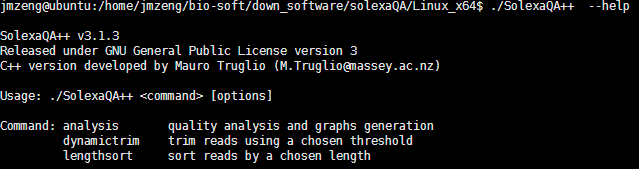
二:命令使用很简单一般使用DynamicTrim与LengthSort.pl就可以了
for id in *fastq
do
echo $id
perl DynamicTrim.pl -454 $id
done
for id in *trimmed
do
echo $id
perl LengthSort.pl $id
done
首先使用DynamicTrim.pl程序,非常耗时间
几个小时完毕之后
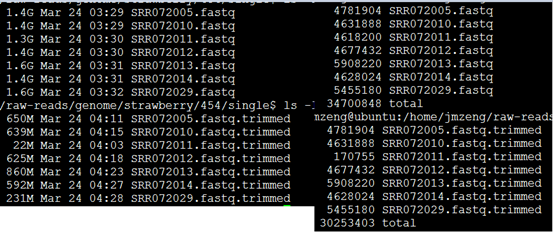
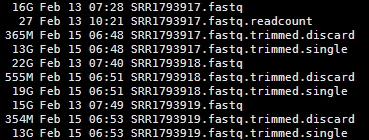
查看,产出文件如下
可以看到丢弃的不多,也就三五百M的
简单查看丢弃的,都是短的。
perl -lne '{print length if $.%4==2}' SRR1793918.fastq.trimmed.discard |head
用这个脚本查看,可知好像都是短于25个碱基的被舍弃掉了,这个参数可以调整的。
接下来就可以用这些数据进行数据分析了