打开官网:http://www.genome.jp/kegg-bin/get_htext?hsa00001+3101
http://www.genome.jp/kegg-bin/get_htext#A1 (这个好像打不开)
可以在里面找到下载链接
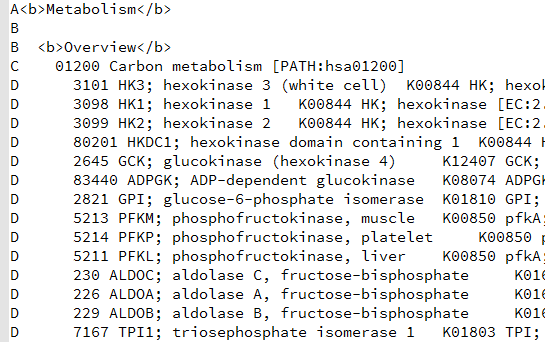
下载得到文本文件,可以看到里面的结构层次非常清楚,
C开头的就是kegg的pathway的ID所在行,D开头的就是属于它的kegg的所有的基因
A,B是kegg的分类,总共是6个大类,42个小类
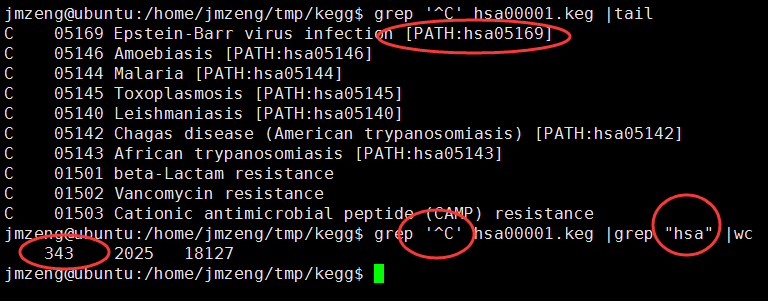
grep ^A hsa00001.keg
A<b>Metabolism</b>
A<b>Genetic Information Processing</b>
A<b>Environmental Information Processing</b>
A<b>Cellular Processes</b>
A<b>Organismal Systems</b>
A<b>Human Diseases</b>
也可以看到,到目前为止(2015年12月8日20:26:57),共有343个kegg的pathway信息啦
接下来我们就把这个信息解析一下:
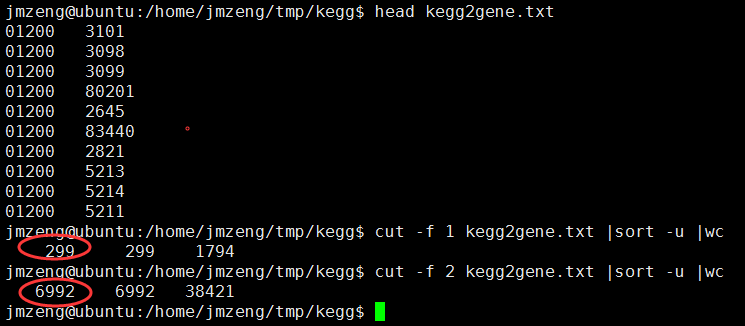
perl -alne '{if(/^C/){/PATH:hsa(\d+)/;$kegg=$1}else{print "$kegg\t$F[1]" if /^D/ and $kegg;}}' hsa00001.keg >kegg2gene.txt
这样就得到了
但是我发现了一个问题,有些通路竟然是没有基因的,我不是很明白为什么?
C 04030 G protein-coupled receptors [BR:hsa04030]
C 01020 Enzyme-linked receptors [BR:hsa01020]
C 04050 Cytokine receptors [BR:hsa04050]
C 03310 Nuclear receptors [BR:hsa03310]
C 04040 Ion channels [BR:hsa04040]
C 04031 GTP-binding proteins [BR:hsa04031]
那我们来看看kegg数据库更新的情况吧。
首先我们看org.Hs.eg.db这个R包里面自带的数据
Date for KEGG data: 2011-Mar15
org.Hs.egPATH has 5869 entrez genes and 229 pathways
2015年八月我用的时候是 6901 entrez genes and 295 pathways
现在是299个通路,6992个基因
所以这个更新其实很缓慢的,所以大家还在用DAVID这种网络工具做kegg的富集分析结果也差不大!