lambda_virus基因组文件是bowtie软件自带的测试数据,共48502个bp,首先我用脚本模拟出它的全打断文件!
perl -alne '{next if /^>/;$a.=$_;}END{$len=length $a;print substr($a.$a,$_,120) foreach 0..$len}' lambda_virus.fa >lamb_virus.120bp
长度均为120bp的片段。
我测序的策略是CTAG碱基重复30次,共加入120个碱基。
对每个120bp片段来说,如果遇到互补碱基就加上,直到120个碱基加完,这样如果比较巧合的话,会有部分碱基能全部加满120bp的,但是如果每个120bp片段的ATCG分布均匀,那么就都应该30bp碱基能被加上。
[perl]
while (<>) {
$seq=$_;$sum=0;
foreach $i (0..120){
$str=substr($seq,$i,2);
if ($str eq "GG"| $str eq "CC"| $str eq "AA"| $str eq "TT"){$sum+=4;}
elsif ($str eq "GT"| $str eq "CG"| $str eq "AC"| $str eq "TA"){$sum+=3;}
elsif ($str eq "GA"|$str eq "CT"| $str eq "AG"| $str eq "TC"){$sum+=2;}
else{$sum+=1;};
#print "$sum\n";
if ($sum>120){print "$i\n";last;}
}
}
[/perl]
perl length.pl lambda_virus.120bp >length.txt
得到结果如下:
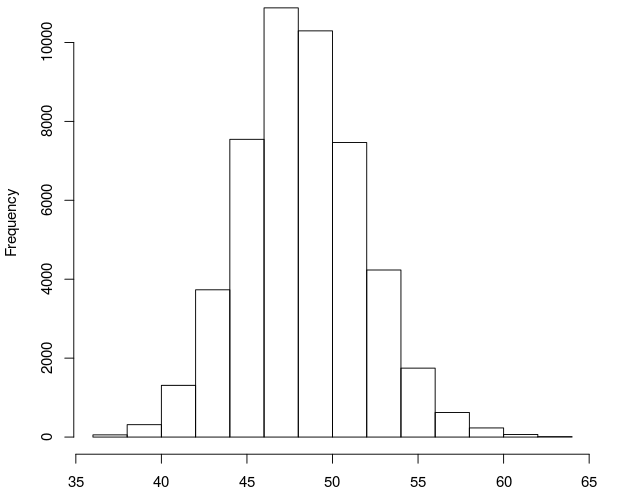
| Length | 36 | 37 | 38 | 39 | 40 | 41 | 42 | 43 | 44 | 45 | 46 | 47 | 48 | 49 | 50 | 51 |
| Count | 2 | 19 | 34 | 110 | 204 | 432 | 878 | 1495 | 2237 | 3202 | 4343 | 5179 | 5697 | 5429 | 4865 | 4214 |
| Length | 52 | 53 | 54 | 55 | 56 | 57 | 58 | 59 | 60 | 61 | 62 | 63 | 64 | |||
| Count | 3249 | 2499 | 1735 | 1090 | 657 | 396 | 228 | 141 | 90 | 48 | 18 | 9 | 3 |
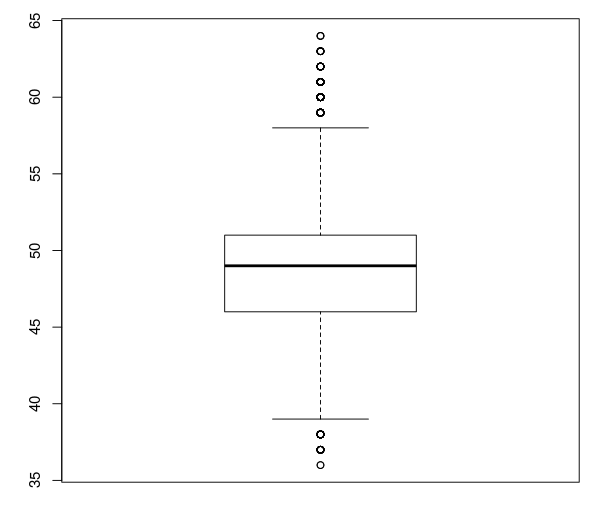
右表可以看出,大部分测序得到碱基长度都集中在46bp到51bp之间
画出箱线图如下
然后我模拟了一个6000bp的基因组,做同样的模拟测序看看评价测序长度分布情况:
| Length | 39 | 40 | 41 | 42 | 43 | 44 | 45 | 46 | 47 | 48 | 49 | 50 | 51 | 52 | 53 | 54 | 55 | 56 | 57 | 58 |
| Count | 9 | 22 | 96 | 207 | 322 | 382 | 479 | 671 | 770 | 714 | 706 | 546 | 424 | 232 | 182 | 100 | 52 | 30 | 14 | 21 |
| Length | 59 | 60 | 61 | |||||||||||||||||
| Count | 15 | 5 | 2 |
可以看出这次的测序片段集中在45到51bp