一般情况下,我们得到了测序reads在基因组的比对情况文件bam格式的,里面的信息非常多,如果我想特定的查看某个基因的情况,那么我们可以选择IGV等可视化工具,但它并不是万能的,因为即使是一个基因,它也会有多个转录本,多个外显子。
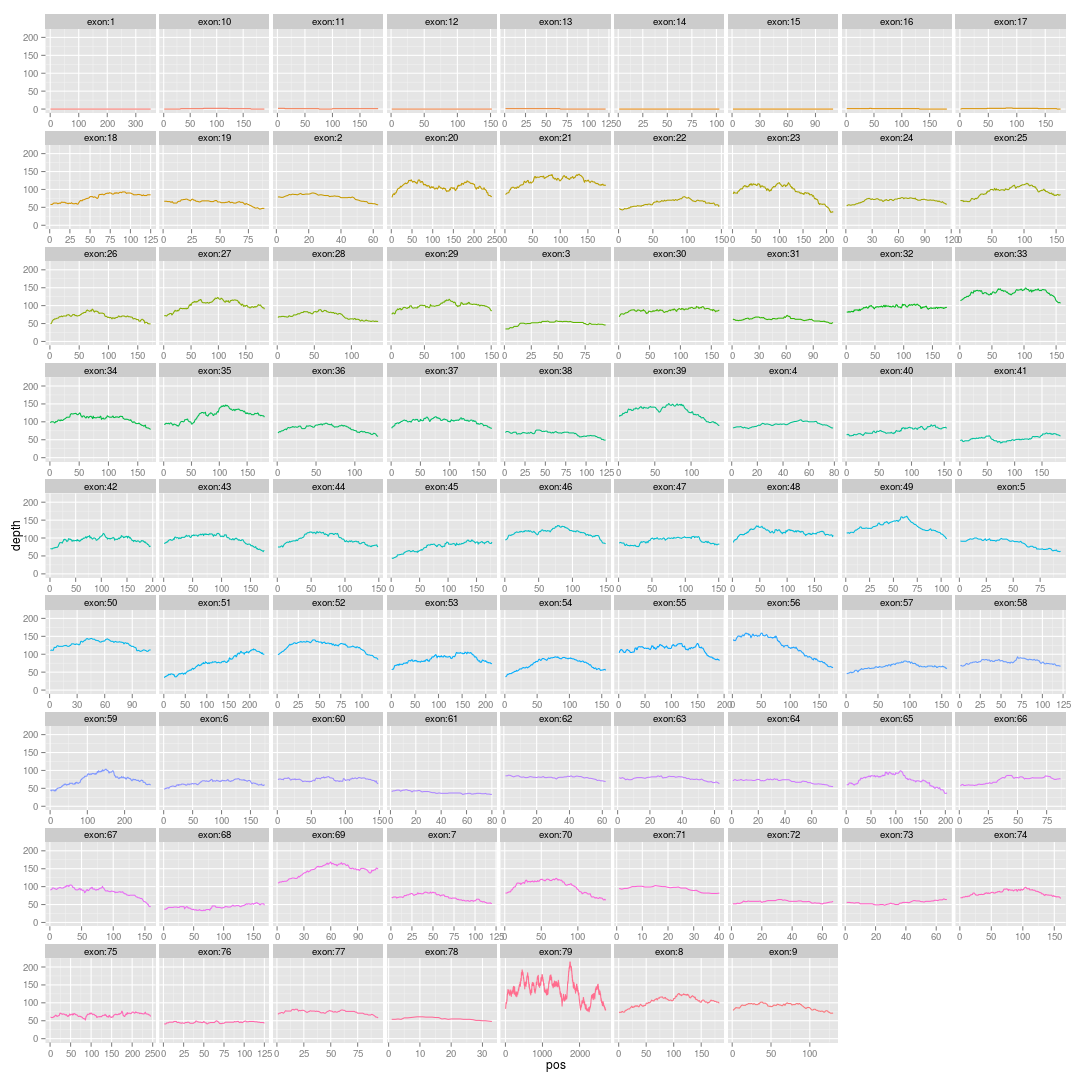
所以,我们可以画它的外显子覆盖图,如下:横坐标是外显子的长度,纵坐标是测序深度,每一个小图都是一个外显子
根据这个图,我们就可以很明显的看出,DMD基因NM_000109转录本的1,10-17号外显子缺失,用IGV一个个的看这些外显子区域,是同样的结果!可能是芯片捕获不到,也可能是样本本身变异,造成的大片段缺失。但是这个图的信息就非常有用!
那么,我们该如何画这样的图呢?
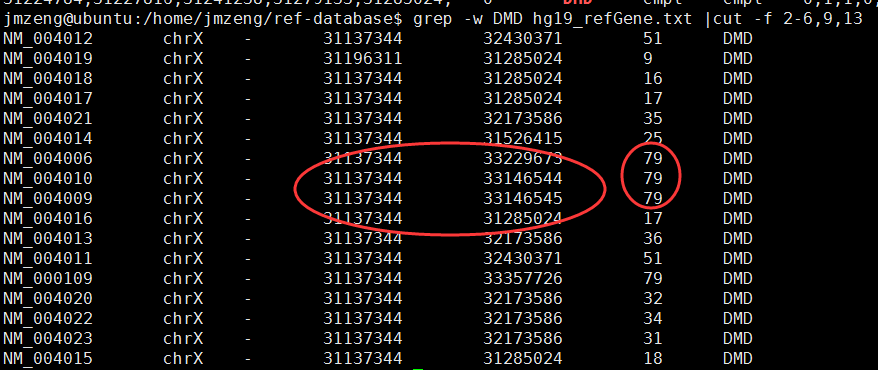
首先,我们需要找到需要探究的基因的全部转录信息,及外显子信息!
在hg19_refGene.txt里面会有,在UCSC里面可以下载,新手可能会比较麻烦,实在不行你去annovar的目录也可以找到!
那么,我们根据这个信息,就可以判断该基因的起始终止位点啦
然后用samtools的depth命令去找这个基因的全部片段的测序深度信息
最后再格式化成下面的三列数据
第一列是该外显子的坐标,从1到该外显子的成都
第二列是该外显子在该坐标的测序深度,通过samtools的depth命令得到
最后一列是该外显子的标记,从exon:79一直倒推到exon:1,因为该基因在染色体的负链,所以外显子顺序是反着的!
1 84 exon:79
2 84 exon:79
3 84 exon:79
4 85 exon:79
5 85 exon:79
6 86 exon:79
7 85 exon:79
8 87 exon:79
9 89 exon:79
10 91 exon:79
11 92 exon:79
12 95 exon:79
13 96 exon:79
14 96 exon:79
15 99 exon:79
16 99 exon:79
17 97 exon:79
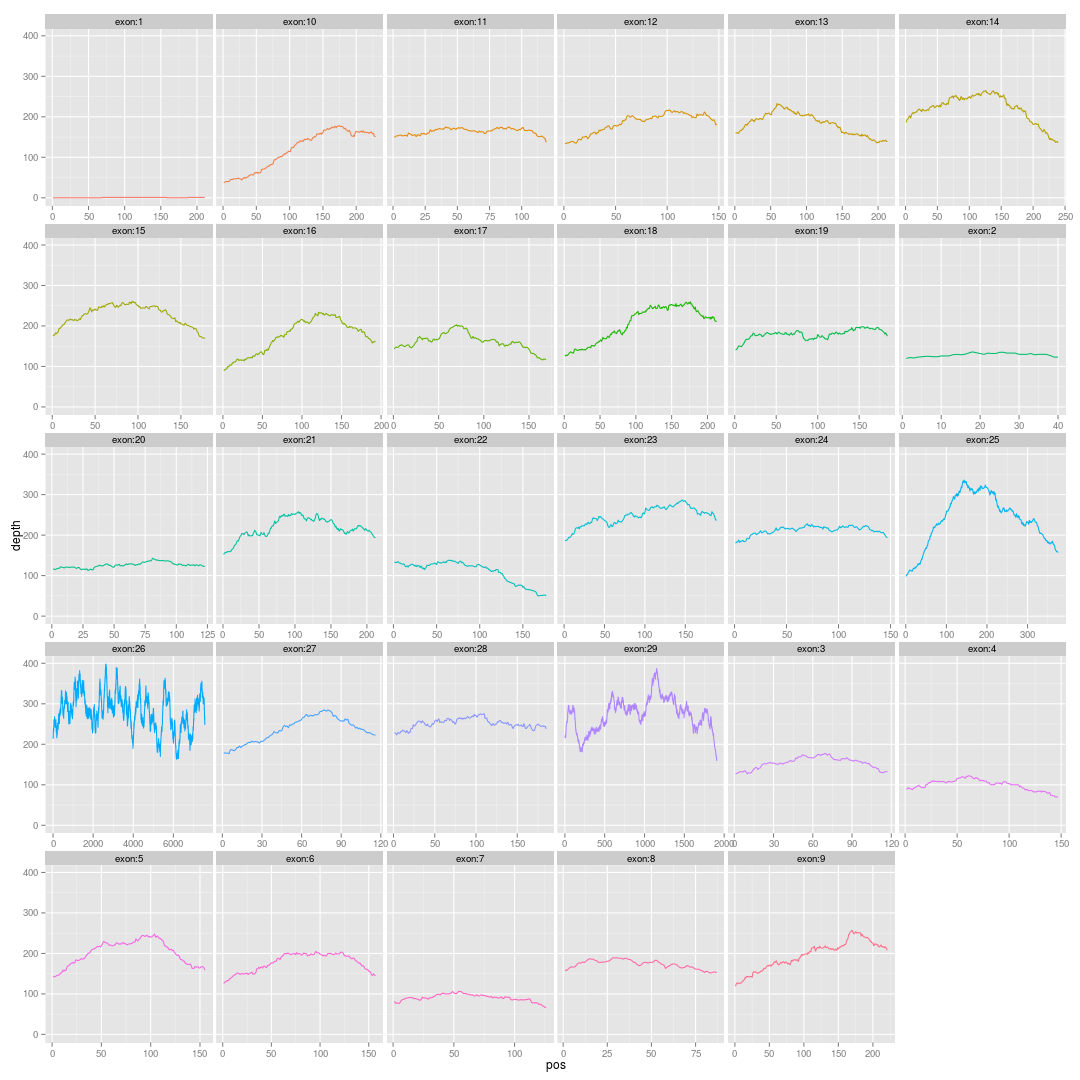
最后根据这个txt文档,用R语言,很容易就画出上面那样的图片了!
这里面的信息量还是蛮大的!