Tag Archives: RNA-seq
跟师妹聊Exome-seq、ChIP-seq、RNA-seq之间的差异
最近学习CHIP-seq的分析流程,略有点心得,也跟以前掌握的WES和RNA-seq做了一些比较,趁跑步的时候跟师妹讨论了一下,正好师妹写了一篇博客来分享这个讨论结果,我也借此机会转载过来,分享给大家,算是借花献佛吧!师妹的博文是用markdown写作,我觉得大家应该直接看她的文章,写得条理清楚:Exome-seq、ChIP-seq、RNA-seq之间的差异 Continue reading
用limma包的voom函数来对RNA-seq数据做差异分析
limma真不愧是最流行的差异分析包,十多年过去了,一直是芯片数据处理的好帮手。
现在又可以支持RNA-seq数据,我赶紧试用了一下!
我下面只讲用法,大家看代码就明白了!
用R语言的DESeq2包来对RNA-seq数据做差异分析
我以前写过DESeq,以及过时了:http://www.bio-info-trainee.com/867.html
正好准备筹集bioconductor中文社区,我写简单讲一下DESeq2这个包如何用!
RNA-seq比对软件HISAT说明书
取代bowtie+tophat进行RNA-seq比对
HISAT全称为Hierarchical Indexing for Spliced Alignment of Transcripts,由约翰霍普金斯大学开发。它取代Bowtie/TopHat程序,能够将RNA-Seq的读取与基因组进行快速比对。这项成果发表在3月9日的《Nature Methods》上。
HISAT利用大量FM索引,以覆盖整个基因组。以人类基因组为例,它需要48,000个索引,每个索引代表~64,000 bp的基因组区域。这些小的索引结合几种比对策略,实现了RNA-Seq读取的高效比对,特别是那些跨越多个外显子的读取。尽管它利用大量索引,但HISAT只需要4.3 GB的内存。这种应用程序支持任何规模的基因组,包括那些超过40亿个碱基的。
HISAT软件可从以下地址获取:http://ccb.jhu.edu/software/hisat/index.shtml。
首先,我们安装这个软件!
Wget http://ccb.jhu.edu/software/hisat/downloads/hisat-0.1.5-beta-source.zip
官网下载的是源码包,需要make一下,make之后目录下面就多了很多程序,绿色的那些都是,看起来是不是很眼熟呀!!!
哈哈,这完全就是bowtie的模拟版本!!!
也可以从github里面下载,wget https://codeload.github.com/infphilo/hisat/zip/master
下载后直接解压即可使用啦。当然这个软件本身也有着详尽的说明书
http://ccb.jhu.edu/software/hisat/manual.shtml
然后就是准备数据,它跟tophat一样的功能。就是把用RNA-seq方法测序得到的fastq文件比对到参考基因组上面,所以就准这两个文件了哦
接下来是运行程序!
说明书上面写着分成两个步骤,构建索引和比对。
这个软件包模仿bowtie自带了一个example数据,而且它的说明书也是针对于那个example来的,我也简单运行一下。
$HISAT_HOME/hisat-build $HISAT_HOME/example/reference/22_20-21M.fa 22_20-21M_hisat
构建索引的命令如上,跟bowtie一样我修改了一下
/home/jmzeng/hoston/RNA-soft/hisat-0.1.5-beta/hisat-build 22_20-21M.fa my_hisat_index
连日志都跟bowtie一模一样,哈哈,可以看到我们的这个参考fasta文件 22_20-21M.fa 就变成索引文件啦,索引还是很多的!
然后就是比对咯,还是跟bowtie一样
$HISAT_HOME/hisat -x 22_20-21M_hisat -U $HISAT_HOME/example/reads/reads_1.fq -S eg1.sam
我的命令是
/home/jmzeng/hoston/RNA-soft/hisat-0.1.5-beta/hisat -x my_hisat_index -U ../reads/reads_1.fq -S reads1.sam
1000 reads; of these:
1000 (100.00%) were unpaired; of these:
0 (0.00%) aligned 0 times
1000 (100.00%) aligned exactly 1 time
0 (0.00%) aligned >1 times
100.00% overall alignment rate
哈哈,到这里。这个软件就运行完毕啦!!!是不是非常简单,只有你会用bowtie,这个就没有问题。当然啦,软件还是有很多细节是需要调整的。我下面就简单讲一个实际的例子哈!
首先,我用了1.5小时把4.6G的小鼠基因组构建了索引
/home/jmzeng/hoston/RNA-soft/hisat-0.1.5-beta/hisat-build Mus_musculus.GRCm38.fa.fa mouse_hisat_index
然后对我的四个测序文件进行比对。
for i in *fq
do
/home/jmzeng/hoston/RNA-soft/hisat-0.1.5-beta/hisat -x /home/jmzeng/hoston/mouse/mouse_hisat_index \
-p 30 -U $i.trimmed.single -S ./hisat_out/${i%.*}.sam
done
它运行的速度的确要比tophat快好多,太可怕的速度!!!!至于是否多消耗了内存我就没有看了
4.6G的小鼠,5G的测序数据,我只用了五个核,居然十分钟就跑完了!
然后听群友说是因为没有加 --known-splicesite-infile <path>这个参数的原因,没有用gtf文件来指导我们的RNA数据的比对,这样是不对的!
需要用下面这个脚本把gtf文件处理一下,然后导入什么那个参数来指导RNA比对。
extract_splice_sites.py genes.gtf > splicesites.txt
但是我报错了,错误很奇怪,没解决,但是我换了个 extract_splice_sites.py 程序,就可以运行啦!之前是HISAT 0.1.5-beta release 2/25/2015里面的python程序,后来我换做了github里面的就可以啦!
/home/jmzeng/hoston/RNA-soft/hisat-master/extract_splice_sites.py Mus_musculus.GRCm38.79.gtf >mouse_splicesites.txt
21192819 reads; of these:
21192819 (100.00%) were unpaired; of these:
14236834 (67.18%) aligned 0 times
5437800 (25.66%) aligned exactly 1 time
1518185 (7.16%) aligned >1 times
感觉没有变化,不知道为什么?
21192819 reads; of these:
21192819 (100.00%) were unpaired; of these:
14236838 (67.18%) aligned 0 times
5437793 (25.66%) aligned exactly 1 time
1518188 (7.16%) aligned >1 times
32.82% overall alignment rate
发表这个软件的文献本身也把这个软件跟其它软件做了详尽的对比
http://www.nature.com/nmeth/journal/v12/n4/full/nmeth.3317.html
| Program | Run time (min) | Memory usage (GB) |
| Run times and memory usage for HISAT and other spliced aligners to align 109 million 101-bp RNA-seq reads from a lung fibroblast data set. We used three CPU cores to run the programs on a Mac Pro with a 3.7 GHz Quad-Core Intel Xeon E5 processor and 64 GB of RAM. | ||
| HISATx1 | 22.7 | 4.3 |
| HISATx2 | 47.7 | 4.3 |
| HISAT | 26.7 | 4.3 |
| STAR | 25 | 28 |
| STARx2 | 50.5 | 28 |
| GSNAP | 291.9 | 20.2 |
| OLego | 989.5 | 3.7 |
| TopHat2 | 1,170 | 4.3 |
参考:http://www.plob.org/2015/03/20/8980.html
http://nextgenseek.com/2015/03/hisat-a-fast-and-memory-lean-rna-seq-aligner/
RNA-seq的比对软件star说明书
类似于tophat的软件
首先当然是下载软件啦!
两个地方可以下载,一个是谷歌code中心,被墙啦,另一个是github,我的最爱。
wget https://codeload.github.com/alexdobin/STAR/zip/master
解压即可使用啦,其中程序在bin目录下面,根据自己的平台调用即可!
然后doc里面还有个pdf的说明文档,写的非常清楚,我也是看着那个文档学的这个软件!
接下来就是准备数据啦!
既然是类似于tophat一样的比对软件,当然是准备参考基因组和测序数据咯,毫无悬念。
然后 该软件也给出了一些测试数据
ftp://ftp2.cshl.edu/gingeraslab/tracks/STARrelease/2.1.4/
然后就是运行程序的命令!
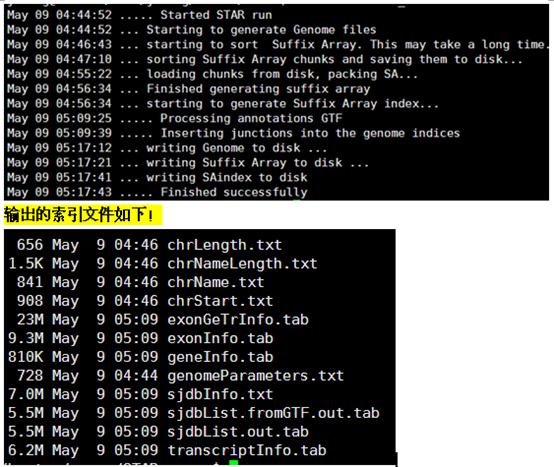
分为两个步骤:首先构建索引,然后比对即可,中间的参数根据具体需要可以细调!
构建索引时候,软件说明书给的例子是
The basic options to generate genome indices are as follows:
--runThreadN NumberOfThreads
--runMode genomeGenerate
--genomeDir /path/to/genomeDir
--genomeFastaFiles /path/to/genome/fasta1 /path/to/genome/fasta2 ...
--sjdbGTFfile /path/to/annotations.gtf
--sjdbOverhang ReadLength-1
我模仿了一下。对我从ensembl ftp里面下载的老鼠基因组构建了索引
/home/jmzeng/hoston/RNA-soft/STAR-master/bin/Linux_x86_64/STAR \
--runThreadN 30 #我的服务器还比较大,可以使用30个CPU \
--runMode genomeGenerate \
--genomeDir /home/jmzeng/hoston/mouse/STAR-mouse #构建好的索引放在这个目录 \
--genomeFastaFiles /home/jmzeng/hoston/mouse/Mus_musculus.GRCm38.fa.fa \
--sjdbGTFfile /home/jmzeng/hoston/mouse/Mus_musculus.GRCm38.79.gtf \
--sjdbOverhang 284 #我的测序数据长短不一,最长的是285bp
当然注释的地方你要删除掉才行呀,因为cpu用的比较多。
算一算时间,对4.6G的小鼠基因组来说,半个小时算是非常快的了!Bowtie2的index要搞两个多小时。
然后就是比对咯。这也是很简单的,软件说明书给的例子是
The basic options to run a mapping job are as follows:
--runThreadN NumberOfThreads
--genomeDir /path/to/genomeDir
--readFilesIn /path/to/read1 [/path/to/read2]
我稍微理解了一下参数,然后写出了自己的命令。
fq=740WT1.fq.trimmed.single
mkdir 740WT1_star
/home/jmzeng/hoston/RNA-soft/STAR-master/bin/Linux_x86_64/STAR \
--runThreadN 20 \
--genomeDir /home/jmzeng/hoston/mouse/STAR-mouse \
--readFilesIn $fq \
--outFileNamePrefix ./740WT1_star/740WT1
如果输出文件需要被cufflinks套装软件继续使用。就需要用一下参数
Cufflinks/Cuffdiff require spliced alignments with XS strand attribute, which STAR will generate with --outSAMstrandField intronMotif option.
还有--outSAMtype参数可以修改输出比对文件格式,可以是sam也可以是bam,可以是sort好的,也可以是不sort的。
最后是输出文件解读咯!
其实没什么好解读的,输出反正就是sam类似的比对文件咯,如果还有其它文件,需要自己好好解读说明书啦。我就不废话了!
值得一提的是,该程序提供了2次map的建议
The basic idea is to run 1st pass of STAR mapping with the usual parameters , then collect the junctions detected in the first pass, and use them as ”annotated” junctions for the 2nd pass mapping.
在对RNA-seq做snp-calling的时候可以用到,尤其是GATK官方还给出了教程,大家可以好好学习学习!
http://www.broadinstitute.org/gatk/guide/article?id=3891
搜索学习其他学者的RNA数据处理流程(包括原始数据、脚本、中间文件)
搜索其他学者的RNA数据处理流程(包括原始数据、脚本、中间文件)
一:原始数据
是谷歌里面无意中搜索到的,是某个物种的RNA数据,不是很大,但是里面有所有的分析流程,非常方便,对原始reads进行了组装,和注释。
http://moana.dnsalias.org/~sgeib/Anth_RNAseq/Run2.1/RawData/
打开网址可以看到raw data的下载链接